0 引 言
随着城市化的快速发展,大量城市河流受到污染,成为黑臭水体。黑臭水体具有异味、水生生物数量减少、河流生态系统结构和功能极度恶化等特点[1],河流黑臭严重威胁了城市居民的健康和生态安全。近年来,国内外已广泛开展利用微生物原位修复技术治理污染河流的研究,取得了较多成果,并证实了利用微生物修复技术治理污染河流是一种既行之有效又保护生态环境,避免二次污染的方法。
微生物法主要是通过投加微生物菌剂等来改善黑臭水质,在我国部分地区实践已取得了一定成效。目前,国内外关于菌剂法的研究主要包括对光合菌[2-3]、酵母菌[4-5]、硝化菌[6-7]和放线菌[8-10]等功能菌群的研究。由于黑臭水体中的污染物成分较为复杂,微生物法对其净化还未达到理想效果。有关研究表明,微生物群落结构越丰富,废水中污染物越易降解[11-14]。水体底泥中含有丰富的微生物,这些微生物是生物转化和污染物去除的主要驱动因素,并决定了废水处理的性能。因此,了解水体底泥中复杂微生物生态学,可以为提高废水处理效率提供理论依据。迄今为止,人们对废水处理中的细菌和古细菌群落已进行了广泛研究[15-16],然而真菌群落结构和功能尚未得到充分证明[17]。真菌是生态系统中普遍存在的微生物和主要参与者[18-19],真菌不仅可以直接降解污染物,还可以促进生态系统的形成和稳定,从而促进降解微生物的发展[20]。最近部分研究表明,真菌种类丰富,显示出巨大的生物技术潜力[21-23],因此,真菌群落的丰富性和作用仍有待探索。
据报道,pH值是影响废水处理中真菌群落结构组成的重要因素之一,低pH可以为废水中真菌的生长提供竞争优势[24]。此外,pH是废水净化处理的重要控制参数之一,它直接影响系统中微生物的生命活力、代谢活力、种类及表面特性,进而对微生物群落结构产生影响。微生物群落结构组成会直接影响污染物处理效率[25]。因此,本文研究投加微生物菌剂后,不同pH对微生物净化黑臭水体的效率和真菌群落结构的影响,以期了解pH与微生物法净化黑臭水体效率之间的关系,评估pH与真菌群落结构之间的关系,为进一步高效治理黑臭水体提供理论和实验依据。
1 材料与方法
1.1 黑臭水体来源
实验上覆水和底泥取自云南省昆明市晋宁县晋城护城河,属GB 3838—2002《地表水环境质量标准》劣Ⅴ类。该河流已严重威胁到沿河居民的生活及身体健康。黑臭水体背景值和GB 3838—2002 Ⅴ类水质标准如表1所示。
1.2 试验方法
1.2.1 pH对菌剂法修复黑臭水体的影响
土著微生物来源于目标水体,较之外来微生物,适应性更好,净化效果更显著。据报道:光合菌、酵母菌、放线菌和硝化菌等菌群在黑臭水体净化中起着重要作用。其中,光合菌、放线菌和硝化菌生长的最适pH为中性或弱碱性条件[26-27],而酵母菌更适合在酸性条件下生长[28]。因此,本研究选取这4类菌群的富集培养物作为微生物菌剂的组成成分,研究投加菌剂法在不同pH条件下对黑臭水体的净化效果及真菌群落结构变化,在试验中控制ρ(DO)为4 mg/L。
表1 黑臭水体水质标准
Table 1 Black odor water background value mg/L

指标ρ(TN)ρ(TP)ρ(COD)ρ(NH3-N)ρ(DO)Ⅴ类水标准20.44022水样背景值5903.537094.90.4
由前期试验可知,在自然状态下(未调节pH,下同),pH在试验第2天时就降至6.0,第3天至试验结束pH基本维持在5.0。在整个试验过程中,净化系统基本处于酸性环境。因此,本研究设置了2个pH净化系统进行试验,分别为:1)菌剂+曝气(pH为5.0),即在整个试验过程中不调节pH;2)菌剂+曝气(pH为7.5),即根据前期研究和实际应用将净化系统pH设置为7.5[29-30],每天调节2次,使净化系统pH在整个试验过程中维持在7.5左右。采用4因素3水平正交试验考察光合菌、酵母菌、硝化菌和放线菌的富集培养物在不同pH条件下对黑臭水体净化效果的影响程度、最佳添加量及比例,每个因素分别包括3个水平,分别为40,60,80 mL/L。试验在摇瓶中进行,分别测定1个试验周期内(6 d,下同)NH3-N、TP和COD的去除率。每组试验进行3次重复,然后计算平均值。由于NH3-N是评价黑臭水体的主要指标,故在本研究中选取NH3-N去除率为主要考察指标。
通过上述正交试验分别确定弱酸性和弱碱性条件下微生物菌剂中各菌群最佳投加量及比例后,分别对2组正交试验结果进行验证。研究以最佳投加量及比例投加菌剂,在1个试验周期(6 d)内,弱酸性和弱碱性条件下微生物对黑臭水体的净化效果进行比较分析。
此外,本研究还设置了曝气试验组,控制ρ(DO)为4 mg/L,试验仅通过曝气处理,研究曝气对黑臭水体的净化效率及其群落结构组成的影响。空白试验组为静置试验,不做任何处理。
1.2.2 试验流程
实验分组编号如表2所示。
表2 实验分组编号
Table 2 Configuration of the experimental groups

编号曝气菌剂pH备注CK空白组T1√曝气组T2√√菌剂+曝气组(pH=5.0)T3√√√菌剂+曝气组(pH=7.5)
注:√表示添加或调节,无√则不添加或不调节。
本研究进行了4组试验,试验周期为6 d,各试验组每天取1次样,测定NH3-N、TP、COD去除率,最后1 d将各试验组样品用无菌离心管进行高速(12000 r/min)离心后,取沉淀保存于-80 ℃冰箱中,送至上海美吉生物医药科技有限公司进行高通量测序分析微生物群落结构。
1.2.3 真菌群落结构多样性分析
取不同处理的各微生物离心样品,在完成基因组DNA抽提后,利用1%琼脂糖凝胶电泳检测抽提的基因组DNA。选用通用引物ITS1F_ITS2R对总DNA样品进行细菌ITS rDNA序列片段扩增,引物序列为:ITS1F:5′-CTTGGTCATTTAGAGGA AGTAA-3′,ITS2R: 5′-GCTGCGTTCTTC ATCGATGC-3′。PCR扩增微生物基因组中ITS rDNA的ITS1F_ITS2R区,PCR反应采用TransStart Fastpfu DNA Polymerase,20 μL反应体系,DNA模板10 ng,反应参数为:95 ℃预变性3 min,然后执行95 ℃变性30 s、55 ℃退火30 s、72 ℃延伸45 s,最后进行72 ℃退火10 min,扩增产物保持在4 ℃条件下。每个样本进行3次重复,将同一样本的PCR产物混合后,用2%琼脂糖凝胶电泳检测。
MiSeq建库及测序工作委托上海美吉生物医药科技有限公司完成。测序数据分析使用I-Sanger云平台完成。
1.3 测定方法
每天定时取样,对分析样品高速离心(8000 r/min) 5 min后,取上清液进行分析检测。NH3-N采用纳氏试剂分光光度法测定[31],COD采用重铬酸钾法测定[31],TP采用钼酸铵分光光度法测定[31],pH值用pH计(Mettle Toledo Delta 320,瑞士)测定,DO采用数字便携式DO测量仪(JPB-607A,中国,杭州)测定。
2 结果与讨论
2.1 pH对菌剂法修复黑臭水体的影响
对净化系统在弱酸性(自然状态)和弱碱性条件下各菌群最佳投加量及比例进行正交试验。正交试验结果如表3和表4所示。
表3 弱酸性条件正交试验结果
Table 3 Orthogonal test results under weakly acidic conditions mL/L
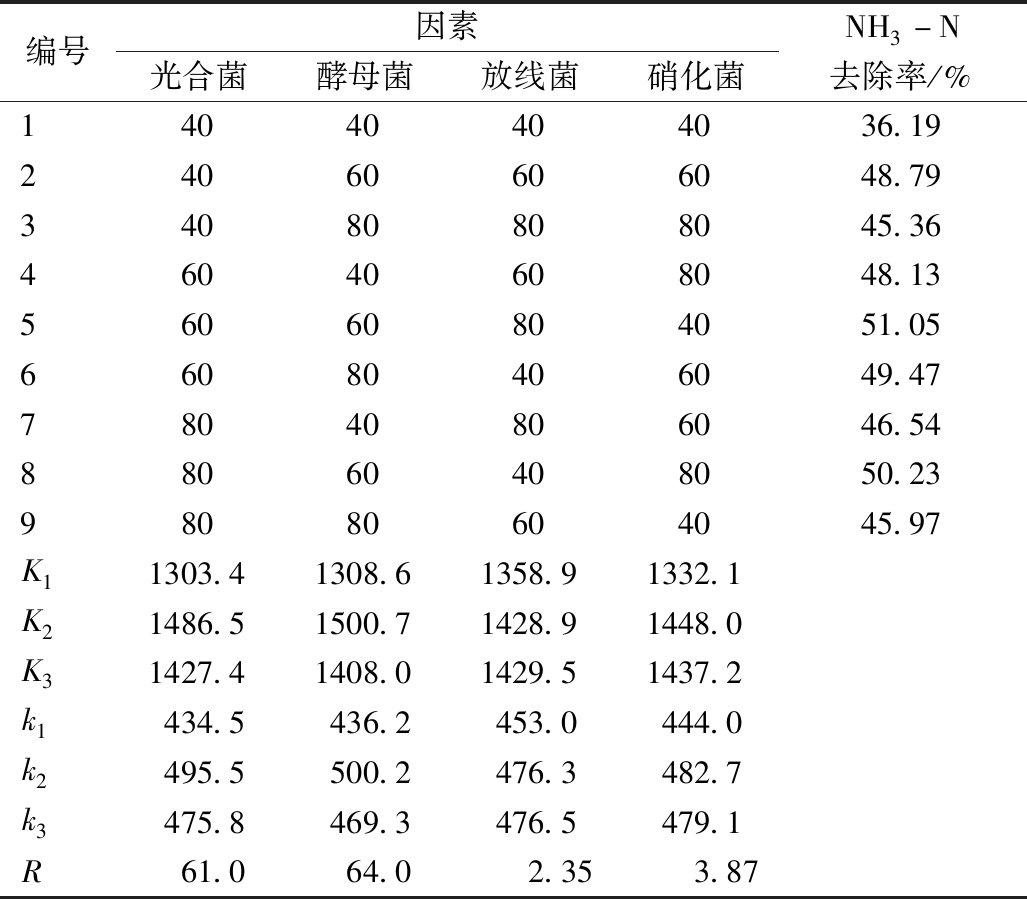
编号因素光合菌酵母菌放线菌硝化菌NH3-N去除率/%14040404036.1924060606048.7934080808045.3646040608048.1356060804051.0566080406049.4778040806046.5488060408050.2398080604045.97K11303.41308.61358.91332.1K21486.51500.71428.91448.0K31427.41408.01429.51437.2k1434.5436.2453.0444.0k2495.5500.2476.3482.7k3475.8469.3476.5479.1R61.064.02.353.87
注:K1、K2、K3分别为某因素1、2、3水平条件下NH3-N去除率之和。k1~k3为K1~K3的平均数,R为极差。下同。
表4 弱碱性条件下正交试验结果
Table 4 Orthogonal test results under weak alkaline conditions
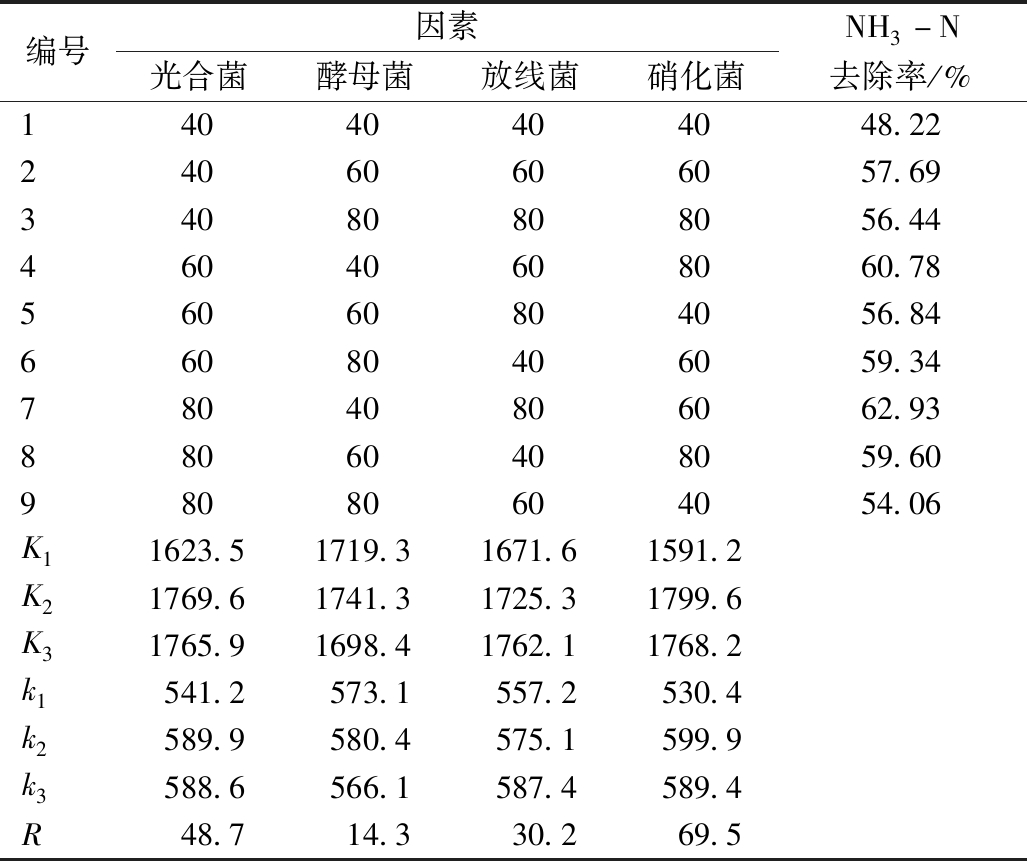
编号因素光合菌酵母菌放线菌硝化菌NH3-N去除率/%14040404048.2224060606057.6934080808056.4446040608060.7856060804056.8466080406059.3478040806062.9388060408059.6098080604054.06K11623.51719.31671.61591.2K21769.61741.31725.31799.6K31765.91698.41762.11768.2k1541.2573.1557.2530.4k2589.9580.4575.1599.9k3588.6566.1587.4589.4R48.714.330.269.5
由表3可知:弱酸性条件下,各菌群对黑臭水NH3-N去除率的影响排序为酵母菌>光合菌>硝化菌>放线菌。最优组合为酵母菌投加量为60 mL/L,光合菌投加量为60 mL/L,硝化菌投加量为60 mL/L,放线菌投加量为80 mL/L,其最佳投加体积比例为3∶3∶3∶4。
由表4可知:弱碱性条件下,各菌群对NH3-N去除率排序为硝化菌>光合菌>放线菌>酵母菌。最优组合为:硝化菌投加量为60 mL/L、光合菌投加量为60 mL/L、放线菌投加量为80 mL/L、酵母菌投加量为60 mL/L,最佳投加体积比为3∶3∶4∶3。
采用t检验法检验两组正交试验对NH3-N脱除率的差异是否显著:tNH3-N=5.08>t0.05=2.12,PNH3-N=0.00<0.05,差异显著。t检验结果表明,pH为7.5时,NH3-N平均去除率显著高于弱酸性条件。这是因为弱碱性环境有利于硝化细菌的生长,强化了硝化反应进行,更有利于去除黑臭水体中的NH3-N。研究表明,维持净化系统为弱碱性环境是提高黑臭水体NH3-N去除率的重要措施之一。
2.2 不同处理对微生物法净化黑臭水体效果的影响
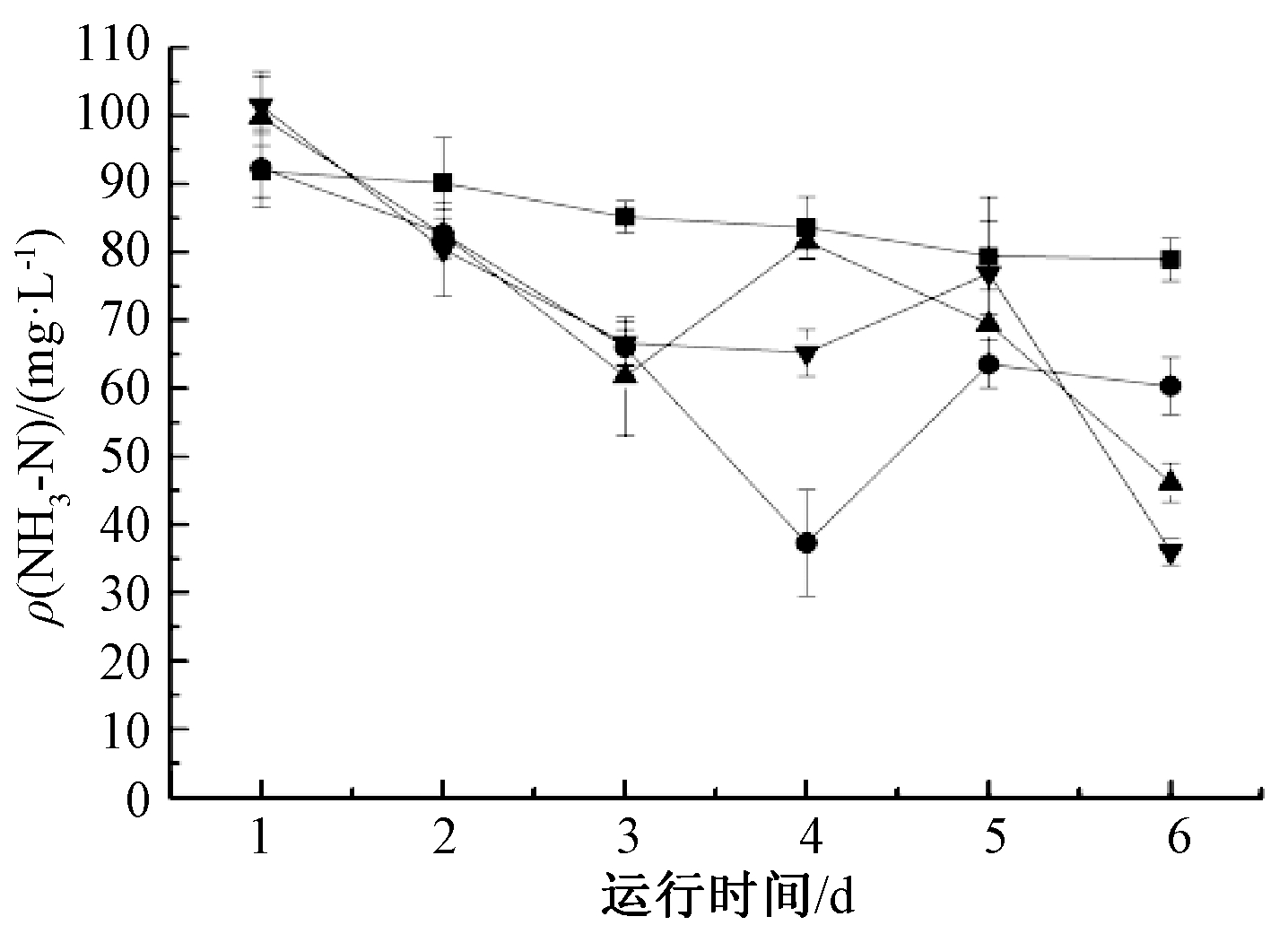
pH对不同污染物去除的影响如图1—3所示。由图1可知:1个试验周期内,CK、T1、T2和T3样品中NH3-N浓度分别下降了12.95,31.98,53.62,65.53 mg/L,去除率分别为14.1%、34.63%、53.72%和63.29%,去除率比较依次为T3>T2>T1>CK。采用t检验法分析T2和T3样品中NH3-N去除率可知,tNH3-N=2.39>t0.05=2.12,PNH3-N=0.048<0.05,差异显著。试验结果表明:投加菌剂对NH3-N的去除有一定促进作用,且弱碱性条件对NH3-N的去除率显著优于弱酸性条件。
![]() —空白;
—空白; ![]() —曝气组;
—曝气组; ![]() —菌剂+曝气组(pH=5.0);
—菌剂+曝气组(pH=5.0); ![]() —菌剂+曝气组(pH=7.5)。
—菌剂+曝气组(pH=7.5)。
图1 pH对NH3-N去除的影响
Fig.1 Effect of pH on NH3-N removal
由图2可知:在1个周期内,CK、T1、T2和T3样品中的ρ(TP)分别降低了0.7,3.34,3.27,3.45 mg/L,对应去除率分别为20.65%、98.53%、95.26%和96.37%。试验结果表明:T2和T3样品中TP去除率变化趋势基本一致,在试验进行6 d后ρ(TP)降至0.2 mg/L,达到GB 3838—2002 Ⅲ类水标准。说明pH对微生物去除TP效率影响不大。

![]() —空白;
—空白; ![]() —曝气组;
—曝气组; ![]() —菌剂+曝气组(pH=5.0);
—菌剂+曝气组(pH=5.0); ![]() —菌剂+曝气组(pH=7.5)。
—菌剂+曝气组(pH=7.5)。
图2 pH对TP去除的影响
Fig.2 Effect of pH on TP removal
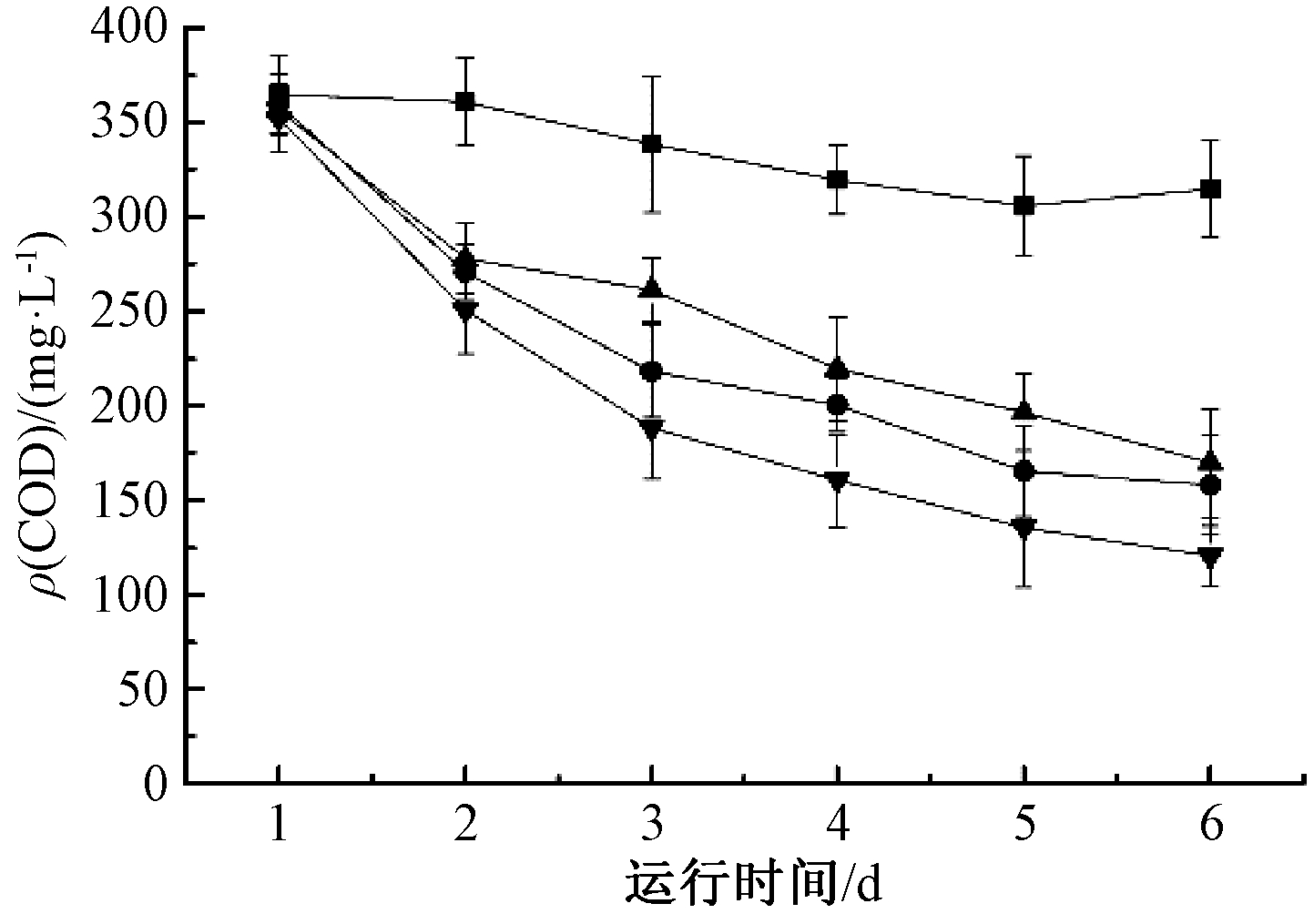
由图3可知:在1个周期内,CK、T1、T2和T3样品中的COD分别下降了50.12,201.68,186.64,231.84 mg/L,去除率分别为13.74%、56.08%,52.39%和65.83%,去除率比较依次为T3>T2>T1>CK。采用t检验法分析T2和T3样品中COD去除率可知:tCOD=2.58>t0.05=2.12,PCOD=0.037<0.05,差异显著。试验结果表明:弱碱性条件更有利于黑臭水体中有机污染物的降解。
![]() —空白;
—空白; ![]() —曝气组;
—曝气组; ![]() —菌剂+曝气组(pH=5.0);
—菌剂+曝气组(pH=5.0); ![]() —菌剂+曝气组(pH=7.5)。
—菌剂+曝气组(pH=7.5)。
图3 pH对COD去除的影响
Fig.3 Effect of pH on COD removal
2.3 微生物群落结构分析
2.3.1 Alpha多样性分析
Alpha多样性可以定量地反映微生物群落的多样性,包括物种丰富度指数和物种多样性指数[32-33]。其测定结果如表5所示。
表5 Alpha多样性指数
Table 5 Alpha diversity index

样本ShannonSimpsonAceChao1Coverage/%CK3.710.1490989799.90T12.150.2774071599.72T21.200.4530523099.89T31.080.5669055599.79
由表5可知:不同处理组中的真菌群落多样性和丰富度差异显著,但Good’s的覆盖率没有观察到显著差异, Good’s的覆盖率(> 99%)表明ITS基因的深度序列足以评估所有样本中的真菌群落多样性[22]。由Shannon和Simpson指数可知,微生物群落多样性从高到低依次为CK>T1>T2>T3;由Ace和Chao1指数可知,微生物群落丰富度从高到低依次为CK>T1>T3>T2,CK中微生物群落多样性和丰富度均高于其他处理组,说明真菌在废水中分布非常广泛。经过不同处理后,α多样性发生下降。在弱酸性条件下,T2中微生物群落丰富度最低,说明弱酸性环境与废水处理过程中真菌生长情况并无直接联系[34]。
2.3.2 各处理间微生物群落结构比较分析
将所有样品分别进行Illumina Miseq高通量测序,将分类单元与Unite(Release 7.0 http://unite.ut.ee/index.php)数据库进行比较,运用Qiime软件[35]剔除疑问数列,并对剩下的序列进行统计与分析,门水平上和属水平上的相对丰度如图4和表6所示。

![]() Basidiomycota;
Basidiomycota; ![]() Rozellomycota;
Rozellomycota; ![]() Zygomycota;
Zygomycota; ![]() Ascomycota。
Ascomycota。
图4 门水平相对丰度
Fig.4 Relative abundance at the phylum level
由图4可知:各试验组优势真菌主要为Ascomycota、Zygomycota、Rozellomycota、Basidiomycota。Ascomycota是所有试验组中最具优势的菌门之一,在各试验组中均占43%以上。据报道[36],Ascomycota是真菌中最大的类群,其物种广泛分布在陆地和水环境中。其是异养型生物,需要有机化合物作为能源,与大多数生物不同,它们能够使用自己的酶来消化植物生物聚合物,如纤维素和木质素。此外,Ascomycota中含有的较多菌属都具有丝状结构,故可以在水力剪切作用下相互缠绕,起到框架作用,有利于细菌附着生长形成好氧颗粒污泥。Zygomycota是所有试验组中的第2大优势菌种,主要分布在土壤中和腐烂的植物体上,具有分解土壤、植物残体和粪便的功能,在碳循环中起着重要作用。
为进一步解析不同处理方式对黑臭水体真菌群落结构的影响,对属水平上相对丰度>1%的优势微生物进行分析比较,结果如表6所示。可知:CK优势真菌主要为:Mortierellales_unclassified(22.51%)、Chaetomiaceae_unclassified(8.23%)、Rozellomycota_unclassified(6.49%)、Aspergillus(6.06%)等,均是废水中常见的真菌[37]。T1优势真菌主要为Mortierellales_unclassified(43.29%)、Scutellinia(8.23%)、Chaetomium(5.63%)等。与CK相比,曝气后Mortierellales_unclassified,Scutellinia和Chaetomium等真菌属相对丰度明显增加,其中,Mortierellales_unclassified为T1最具优势的真菌,这与Hu的研究一致[38]。研究表明:Mortierellales_unclassified与放线菌一样可以有效地降解几丁质,其中的一些物种能够通过使用木聚糖酶将半纤维素降解为糖,为它们的生长提供糖分[39]。Scutellinia已被证明是废水处理系统中的主要真菌[37]。Chaetomium有利于工业纺织废水的脱色和生物降解,其在废水、废物和污染土壤的净化处理方面具有很大的潜力[40]。
由表6还可以看出:T2优势真菌主要为Candida(51.52%)、Mortierellales_unclassified(7.79%)、Rozellomycota_unclassified(7.79%)和Kazachstania(6.49%)等。T3优势真菌主要为Mortierellales_unclassified(45.45%)、Scutellinia(6.93%)、Candida(6.06%)、Rozellomycota_unclassified(6.06%)和Kazachstania(4.33%)等。与T1相比,T2、T3中Candida、Kazachstania和Penicillium等真菌明显增加并成为新的优势属。Candida是一种酵母属,其在投加菌剂的试验组中相对丰度较高,尤其是在酸性条件下,这可能与微生物菌剂中含有酵母菌有关。据报道[41],Candida广泛存在于废水处理厂,有利于铬去除和苯酚降解[42-43],且有研究证明Candida具有高效降解氨氮和淀粉的能力[44]。Kazachstania具有较高的产胞外多糖的能力[45]。Penicillium具有降低乳制品废水中脂质含量的作用[46,47],以及协助可溶性蛋白质和糖类等有机污染物的降解和提高污泥沉降性能。T2与T3相比,Candida、Kazachstania和Trichoderma等真菌属明显增加。Trichoderma在废水、淤泥的生物降解和转化方面具有重要作用[48],并具有合成拮抗化合物(蛋白质、酶和抗生素)和微量营养素(维生素、激素和矿物质)的能力[49]。Mortierellales_unclassified和Scutellinia等菌属明显减少。
表6 属水平相对丰度
Table 6 Relative abundance at genus level %

物种CKT1T2T3Mortierellales_unclassified22.5143.297.7945.45Candida0.430.0051.526.06Rozellomycota_unclassified6.493.907.796.06Scutellinia1.738.230.436.93Chaetomiaceae_unclassified8.233.462.163.46Aspergillus6.062.161.730.87Kazachstania0.000.006.494.33Chaetomium3.035.630.000.43Trichosporon2.160.433.460.87Trichoderma2.602.160.431.30Gibellulopsis3.030.870.871.73Pseudeurotium2.601.300.431.73Guehomyces3.033.030.000.00Arthrographis2.601.300.430.87Rhodotorula2.601.730.430.43Penicillium0.430.871.731.73Monographella2.161.300.430.87Nectriaceae_unclassified0.432.160.431.30Cephaliophora3.460.430.000.00Mortierella1.731.730.430.00Ascomycota_unclassified1.731.300.430.43Microascaceae_unclassified1.730.870.430.87Pseudallescheria1.300.870.870.87Scedosporium0.431.301.300.43Fusarium1.730.000.870.43Pyrenochaetopsis2.160.000.000.43Sporobolomyces0.000.871.730.00Microascales_unclassified0.430.001.300.43Davidiellaceae_unclassified1.300.000.000.43Pseudaleuria0.000.000.431.30Sterigmatomyces1.300.000.000.00Pezizales_unclassified1.300.000.000.00Pleurostomophora1.300.000.000.00others9.9610.825.639.96
综上,本研究发现优势真菌群落在不同处理方式中具有高度动态性。据报道[50],与细菌相比,真菌具有能够在低pH、低氮含量的条件下生长的优势。部分真菌在污泥处理中具有以下显著优点:1)真菌具有降解多种有机物的能力,同时可以提高污泥的沉淀性能;2)能够减少病原体和去除有毒化合物;3)真菌处理废水能耗较低,氧气供应需求约为细菌需氧量的1/3等。然而,真菌在污泥处理中用于改善污泥沉降、脱水和降解的作用尚未确定。目前,大多数关于真菌处理废水研究处于小试阶段。因此,未来工业过程应用需在广泛的基本实验室工作基础上,进行中试规模研究。此外,在后期研究中可以通过各种操作条件(如溶解氧、pH、温度、营养物质、操作条件和培养时间)的优化,选择优势或功能真菌与其他微生物结合以提高降解能力,确保生态系统(复杂的真菌混合物)的稳定性、适应性和表达性[38],在特定条件下实现微生物处理废水的优异性能[49]。
3 结 论
1)在曝气辅助条件下,投加菌剂并维持净化系统为弱碱性条件,微生物对NH3-N和COD去除率均明显高于弱酸性条件。但不同pH条件下,微生物对TP去除率基本一致。
2)高通量测序分析发现,不同处理组中微生物群落多样性和丰富度差异显著。门水平上,各试验组优势真菌主要为Ascomycota、Zygomycota、Rozellomycota、Basidiomycota;属水平上,不同pH条件下真菌群落结构及优势菌种发生了改变。与本研究前期试验关于废水处理过程中细菌的群落变化相比,真菌群落表现出更高的聚集性,并且受到操作条件和环境变量pH的影响较大。
[1] Defu H, Chen R R, Zhu E H, et al. Toxicity bioassays for water from black-odor rivers in wenzhou, China [J]. Environmental Science and Pollution Research, 2015,22(3):1731-1741.
[2] Lu H, Zhang G, Dai X, et al. A novel wastewater treatment and biomass cultivation system combining photosynthetic bacteria and membrane bioreactor technology [J]. Desalination, 2013,322:176-181.
[3] Kaewsuk J, Thorasampan W, Thanuttamavong M, et al. Kinetic development and evaluation of membrane sequencing batch reactor (msbr) with mixed cultures photosynthetic bacteria for dairy wastewater treatment [J]. Journal of Environmental Management, 2010,91(5):1161-1168.
[4] Zak S. The use of fenton?S system in the yeast industry wastewater treatment [J]. Environmental Technology Letters, 2005,26(1):11-19.
[5] Shen Q H, Wang X J, Li X J. Research and application progress of yeast in wastewater treatment [J]. Applied Mechanics and Materials, 2014,587-589:669.
[6] Motesharezadeh B, Arasteh A, Pourbabaee A A, et al. The effect of zeolite and nitrifying bacteria on remediation of nitrogenous wastewater substances derived fromcarp breeding farm [J] International Journal of Environmental Research, 2015,9(2):553-560.
[7] Inui T, Tanaka Y, Okayas Y, et al. Application of toxicity monitor using nitrifying bacteria biosensor to sewerage systems [J]. Water Science and Technology, 2002,45(4/5):271-278.
[8] Herman A, Kappler J W, Marrack P, et al. Isolation and preliminary identification of actinomycetes isolated from a wastewater treatment plant and capable of growing on methyl ethyl ketone as a sole source of carbon and energy[J]. Desalination and Water Treatment, 2016, 57(26): 12108-12117.
[9] Soler A, Garcíahernández J, Zornoza A, et al. Diversity of culturable nocardioform actinomycetes from wastewater treatment plants in spain and their role in the biodegradability of aromatic compounds [J]. Environmental Technology, 2017,39(4): 1-10.
[10] Bezak-Mazur E, Stoińska R, Rajca J, et al. Effect of the presence of actinomycetes in the activated sludge on the quality of the treated wastewater [C]∥EES Web of Conferences, 2017,17:00007.
[11] Meerbergen K, Geel M V, Waud M, et al. Assessing the composition of microbial communities in textile wastewater treatment plants in comparison with municipal wastewater treatment plants [J]. Microbiologyopen, 2017,6(1):1-13.
[12] B aszczyk K,
aszczyk K, ![]() T. Microbial diversity of sewage sludge [J]. IEEE, 2013,7(2):461-466.
T. Microbial diversity of sewage sludge [J]. IEEE, 2013,7(2):461-466.
[13] Fierer N, Jackson R B. The diversity and biogeography of soil bacterial communities [J]. PNAS, 2006,103(3):626-631.
[14] Farzadkia M, Bazrafshan E. Lime stabilization of waste activated sludge [J]. Health Scope. 2014,3(1): e16035.
[15] Fredriksson N J, Hermansson M, Wilén B M. Diversity and dynamics of archaea in an activated sludge wastewater treatment plant [J]. BMC Microbiology,2012,12(1):140.
[16] Hashimoto K, Matsuda M, Inoue D, et al. Bacterial community dynamics in a full-scale municipal wastewater treatment plant employing conventional activated sludge process [J]. Journal of Bioscience and Bioengineering, 2014,118(1): 64-71.
[17] Niazi S, Hassanvand M S, Mahvi A H, et al. Assessment of bioaerosol contamination (bacteria and fungi) in the largest urban wastewater treatment plant in the middle east [J]. Environmental Science and Pollution Research International, 2015,22(20): 16014-16021.
[18] Dighton J . Nutrient cycling by saprotrophic fungi in terrestrial habitats[M]∥The Mycota Ⅳ Environmental and Microbial Relationships (2nd Ed.). Springer Berlin Heidelberg, 2007.
[19] Ramoni J, Seiboth B. 6 degradation of plant cell wall polymers by fungi [M]∥Environmental and Uicrobial Pelationship. Springer International Publishing, 2016.
[20] Liu J, Li J, Tao Y, et al. Analysis of bacterial, fungal and archaeal populations from a municipal wastewater treatment plant developing an innovative aerobic granular sludge process [J]. World Journal of Microbiology and Biotechnology, 2017,33(1):14-21.
[21] Evans T N. Estimating biodiversity of fungi in activated sludge communities using culture-independent methods [J]. Microbial Ecology, 2012,63(4):773-786.
[22] Maza-Márquez P, Vilchez-Vargas R, Kerckhof F M, et al. Community structure, population dynamics and diversity of fungi in a full-scale membrane bioreactor (mbr) for urban wastewater treatment [J]. Water Research, 2016,105:507- 519.
[23] Yang Q, Angly F E, Wang Z, et al. Wastewater treatment systems harbor specific and diverse yeast communities [J]. Biochemical Engineering Journal, 2011,58(1):168-176.
[24] Zheng S, Sun J, Han H. Effect of dissolved oxygen changes on activated sludge fungal bulking during lab-scale treatment of acidic industrial wastewater[J]. Environmental Science and Technology, 2011, 45(20):8928-8934.
[25] Guo Y, Peng J, Song Y, et al. Influence of temperature on the pollutant removal efficiency and the microbial community of the anaerobic baffled reactor [J]. Acta Scientiae Circumstantiae, 2012,32(7):1542-1548.
[26] 王琳. 一株沼泽红假单胞菌VOTO1-G的分离鉴定及应用研究[J]. 卫生研究, 2012,41(6):938-942.
[27] 《环境科学大辞典》编委会. 环境科学大辞典(修订版) [M]. 北京:中国环境科学出版社, 2008.
[28] Camargo F P, De A M, Tonello P S, et al. Characterization of biosurfactant from yeast using residual soybean oil under acidic conditions and their use in metal removal processes [J]. FEMS Microbiology Letters, 2018,365(10):1-8.
[29] 尚然, 岳秀丽, 马放,等. 好氧反硝化菌的反硝化特性及pH值和温度影响性研究[C]∥中国微生物生态学年会, 2008.
[30] 史旭东, 荣宏伟, 张朝升, 等. pH对SBR工艺亚硝酸型SND及过程控制的影响 [J]. 环境工程, 2012,29(13):61-66.
[31] Zheng B, Zhang Q, Zhang Y, et al. Heterogeneous chemistry: A mechanism missing in current models to explain secondary inorganic aerosol formation during the january 2013 haze episode in north china [J]. Atmospheric Chemistry and Physics, 2015,14(15):2031-2049.
[32] Li B, Zhang X, Guo F, et al. Characterization of tetracycline resistant bacterial community in saline activated sludge using batch stress incubation with high-throughput sequencing analysis [J]. Water Research, 2013,47(13):4207-4216.
[33] Lundberg D S, Yourstone S, Mieczkowski P, et al. Practical innovations for high-throughput amplicon sequencing [J]. Nature Methods, 2013,10(10):999-1022.
[34] Maza-Márquez P, Vílchez-Vargas R, González-Martínez A, et al. Assessing the abundance of fungal populations in a full-scale membrane bioreactor (MBR) treating urban wastewater by using quantitative PCR (qPCR)[J]. Journal of Environmental Management, 2018, 223:1-8.
[35] Caporaso J G, Kuczynski J, Stombaugh J, et al. Qiime allows analysis of high-throughput community sequencing data [J]. Nat Methods, 2010,7(5):335-336.
[36] Schoch CL, Gi-Ho S, Francesc L G, et al. The ascomycota tree of life: A phylum-wide phylogeny clarifies the origin and evolution of fundamental reproductive and ecological traits [J]. Syst Biol, 2009,58(2):224-239.
[37] Zhang H, Feng J, Chen S, et al. Disentangling the drivers of diversity and distribution of fungal community composition in wastewater treatment plants across spatial scales[J]. Frontiers in Microbiology, 2018, 9:1291.
[38] Hu M, Wang X, Wen X, et al. Microbial community structures in different wastewater treatment plants as revealed by 454-pyrosequencing analysis. Bioresour technol [J]. Bioresource Technology, 2012,117(10):72-79.
[39] Dix N J, Webster J. Fungal Ecology [M]. London: Chapman & Hall, 1994.
[40] Manai I, Miladi B, El M A, et al. Industrial textile effluent decolourization in stirred and static batch cultures of a new fungal strain chaetomium globosum ima1 kj472923 [J]. Journal of Environmental Management, 2016,170:8-14.
[41] Wei Z, Liu Y, Feng K, et al. The divergence between fungal and bacterial communities in seasonal and spatial variations of wastewater treatment plants [J]. Science of the Total Environment, 2018, 628/629:969-978.
[42] Adav S S, Chen M Y, Lee D J, et al. Degradation of phenol by aerobic granules and isolated yeast candida tropicalis [J]. Biotechnology and Bioprocess Engineering, 2010,96(5): 844-852.
[43] Ye J, Hua Y, Mai B, et al. Biosorption of chromium from aqueous solution and electroplating wastewater using mixture of candida lipolytica and dewatered sewage sludge [J]. Bioresource Technology, 2010,101(11):3893-3902.
[44] Sawai T, Hehre E J. A novel amylase (candida transglucosyl-amylase) that catalyzes glucosyl transfer from starch and dextrins [J]. Journal of Biological Chemistry, 1962,237(7):2047-2052.
[45] Zhou J, Liu X, Jiang H, et al. Analysis of the microflora in tibetan kefir grains using denaturing gradient gel electrophoresis [J]. Food Microbiology, 2009,26(8):770-775.
[46] Leal M C M R, Freire D M G, Cammarota M C, et al. Effect of enzymatic hydrolysis on anaerobic treatment of dairy wastewater [J]. Process Biochemistry, 2006,41(5):1173-1178.
[47] Duarte J G, Silva L L S, Freire D M G, et al. Enzymatic hydrolysis and anaerobic biological treatment of fish industry effluent: evaluation of the mesophilic and thermophilic conditions [J]. Renewable Energy, 2015,83:455-462.
[48] Molla A H, Fakhru′L-Razi A, Abd-Aziz S, et al. A potential resource for bioconversion of domestic wastewater sludge [J]. Bioresource Technology, 2002,85(3):263-272.
[49] More T T, Yan S, Tyagi R D, et al. Potential use of filamentous fungi for wastewater sludge treatment [J]. Bioresource Technology, 2010,101(20):7691-7700.
[50] Sladka A, Ottová V. The most common fungi in biological treatment plants [J]. Hydrobiologia, 1968,31(3/4):350-362.