0 引 言
通过粪便、尿液等排泄物[1]排出生物体的抗生素(antibiotics)作为特殊的环境选择压力,使携带抗生素抗性基因(antibiotic resistance genes, ARGs)的微生物成为优势物种,来自内在抗性与外源输入[2]的ARGs在人类、动物、生态环境间不断复制、传递和循环[3],被ARGs感染后的细胞将使相应的抗生素失效[4],给人类健康和生态安全带来重大威胁。根据存在形式,ARGs分为胞内ARGs和胞外ARGs,目前对胞外DNA所携带ARGs的研究较少[5]。ARGs在不同细菌间通过水平基因转移(horizontal gene transfer, HGT)的方式传播扩散[6],细菌从环境中直接吸收胞外DNA而获得新基因的方式正是利用“转化”手段进行传播扩散。因此,胞外ARGs的抗性风险不容小觑。
在污染治理体系中,污水处理系统(wastewater treatment plants, WWTPs)尤为重要,是ARGs重要的“汇”和“源”:一方面,它在汇聚各类污废水时汇集了大量不同来源的ARGs[7];另一方面,污水处理系统是ARGs诱导和迁移的场所,是ARGs进入自然生态环境前的最后一道屏障[8]。生活污水处理系统所处理的生活污水含有大量的易腐化有机物和病原微生物,来源广、总量大,生活污水处理系统改善了生态环境,是建设“美丽中国”的必要基础设施,与人类生活密切相关,是污水处理系统的关键一环。因此,为了识别ARGs在生活污水处理系统中的潜在抗性风险,保护人类健康及生态安全,对生活污水处理系统全流程(包括污水、污泥处理流程)中的胞内、胞外ARGs开展研究十分必要。
本研究选取河北省某A2/O生活污水处理系统为研究对象,于污水、污泥处理工艺沿程各单元采集污水、污泥样品。采用普通PCR技术和实时荧光定量PCR技术,对污水、污泥处理工艺沿程各单元样品的胞内、胞外ARGs进行试验分析,揭示生活污水处理系统中胞内、胞外ARGs的分布变化与去除效率。为降低乃至消除污水处理系统中ARGs的抗性风险、制定关于ARGs污染的排放标准提供一定的理论依据。
1 材料与方法
1.1 污水处理系统简介
选取河北省某A2/O生活污水处理系统(编号为YJ)为研究对象,其日处理生活污水(100%来源)约5万t,主体工艺为A2/O工艺,处理后污水达到GB 18918—2002《城镇污水处理厂污染物排放标准》一级A标准。整体工艺分为预处理阶段、生化处理阶段、深度处理阶段。对污水处理系统进行基本信息参数的调研、汇总如表1所示,具体污水、污泥处理工艺流程及采样点分布如图1所示。
表1 YJ污水处理系统基本信息参数
Table 1 Basic information of the YJ wastewater treatment plant

污水厂基本信息名称信息参数详情主体工艺A2/O污水处理量约50000 m3/d进水组成100% 生活污水进水ρ(COD)200~300 mg/L出水ρ(COD)<50 mg/L(符合GB 18918—2002一级A排放标准)曝气池污泥龄20 d絮凝池絮凝剂PAM、PAC尾水储池消毒剂ClO2脱水外运污泥含水率80%~90%(加PAM助脱水)脱水外运污泥处置方式卫生填埋处置
1.2 污水、污泥样品采集
采样时间为2018年7月4日,污水、污泥处理流程采样点见图1(编号1—15)。采样点具体涉及总进水,旋流沉砂池出水,A2/O生化池厌氧区、缺氧区、好氧区泥水混合液,二沉池出水,絮凝沉淀池出水,纤维转盘滤池出水,ClO2消毒池出水,二沉池污泥回流液,污泥浓缩池上清液,脱水机房冲洗液(含/不含污泥脱水滤液),泥区废液总回流液,脱水前污泥和脱水后污泥。
污水样品采集及保存使用5 L聚乙烯桶,每个样点采集2次,所用器皿在使用前经过10%漂白剂浸泡以去除DNA残留[9],而后依次使用自来水、去离子水、Millipore超纯水冲洗干净;污泥样品采集存放于洁净无菌的封口塑料袋中,脱水前、后污泥各装2袋。所有水、泥样品采集后于3 h内冷藏运输回实验室,污水样品于4 ℃冰箱保存,污泥样品于-80 ℃冻存,并立即进行各样品的水质因子分析、预处理、胞内外DNA的提取以及胞内外ARGs的分析。样品水质因子(pH、COD、NH3-N)检测结果如表2所示。
1.3 样品胞内基因组DNA的提取
1.3.1 胞内DNA提取
污水样品的胞内DNA预处理采用0.22 μm混合纤维素酯滤膜(Millipore, 德国)抽滤截留的方法,并记录抽滤所用污水样品体积,将载有样品的滤膜用洁净无菌的剪刀剪碎,转移至洁净的收集管中;污泥样品的胞内DNA预处理为先使用冻干机冻干污泥样品72~96 h,保证污泥样品完全干燥后,称取适量冻干污泥样品转移到洁净无菌的收集管中。随后两者均立即使用PowerSoil DNA Isolation Kit(Mo Bio, 美国)试剂盒进行样品胞内DNA的提取,每个点位的污水、污泥胞内DNA样品均设置2组平行。提取完成后,使用Nanodrop 2000(Thermo Fisher,美国)对样品胞内DNA的浓度和纯度进行检测。
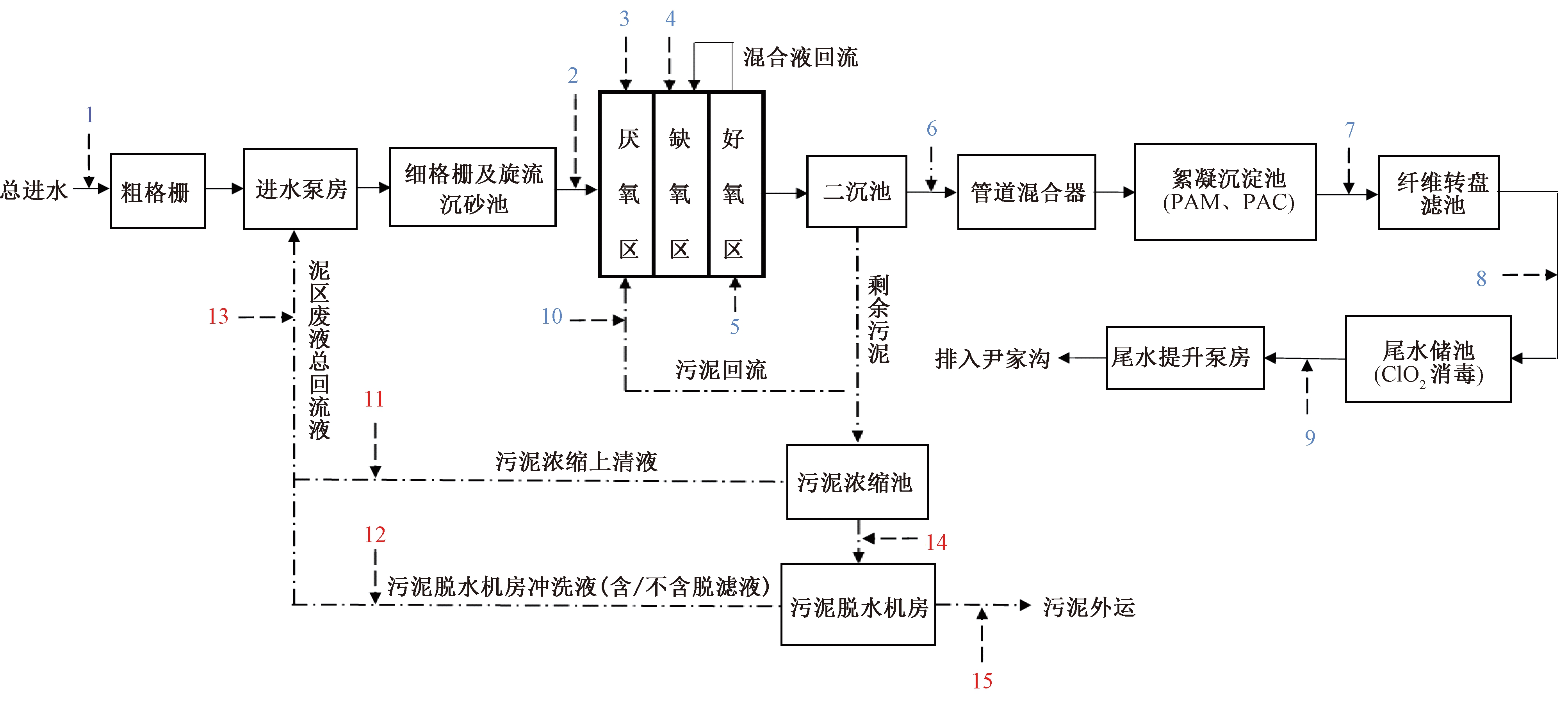
![]() 污水处理流程;
污水处理流程;![]() 污泥处理流程;采样点 1—15,蓝色为污水处理流程采样点,红色为污泥处理流程采样点。
污泥处理流程;采样点 1—15,蓝色为污水处理流程采样点,红色为污泥处理流程采样点。
图1 YJ污水处理系统工艺流程及采样点分布示意
Fig.1 Treatment process of the YJ wastewater treatment plant and the sampling sites’arrangement
表2 样品水质情况
Table 2 Water quality idexes of the samples mg/L(pH除外)

样品编号样品名称pHρ(COD)ρ(NH3-N)1总进水7.13238.33±8.1629.050±1.3442旋流沉砂池出水7.21198.00±2.6831.600±0.1413A2/O生化池厌氧区泥水混合液6.8671.33±1.6319.500±0.4244A2/O生化池缺氧区泥水混合液6.7860.00±2.538.750±2.6165A2/O生化池好氧区泥水混合液6.7890.00±2.199.695±0.7426二沉池出水7.4749.67±0.821.350±0.0147絮凝沉淀池出水7.6729.00±1.670.950±0.0148纤维转盘滤池出水7.836.29±2.691.320±0.0009ClO2消毒池出水7.6839.33±1.031.290±0.12710二沉池污泥回流液6.6778.00±1.793.850±0.12711污泥浓缩池上清液7.3150.00±1.261.030±0.01412脱水机房冲洗液(含污泥脱水滤液)7.29174.17±2.042.200±0.00012′脱水机房冲洗液(不含污泥脱水滤液)7.2651.67±4.087.600±0.00013泥区废液总回流液6.8287.50±5.243.100±0.566
1.3.2 胞外DNA提取
每个污水样品(除脱水前、后污泥)取1 L样品于4 ℃下静置30 min,使水体悬浮物基本沉于水体底部。转移上清液并使用0.22 μm滤膜进行抽滤,去除水体中残留的悬浮物和细胞。除去细胞等杂质的0.22 μm滤膜滤出液采用乙醇沉淀法进行胞外DNA的收集[5]:将726 mL无水乙醇(分析纯)和33 mL的3 mol/L乙酸钠(分析纯)加入装有330 mL滤出液的蓝盖瓶中,密封混匀数次;将混合溶液置于-20 ℃冷藏12 h以上,随后将其置于高速冷冻离心机进行10000 g离心10 min,弃除上清液;收集到的沉淀转移至洁净无菌的收集管中,提取胞外DNA的操作、DNA浓度和纯度的检测操作同上。每个点位的污水胞外DNA样品均设置2组平行。
1.4 普通PCR定性检测
为了使所选目标待测基因具有一定的代表性,尽可能选择近年来研究检出频率及丰度较高、不同种类且具有不同耐药作用机制的ARGs(如tetC属于“外输泵”类机制,sulII属于“目标分流”类机制,ermB属于“目标改性”类机制等)进行研究,同时近年来有研究表明,Ⅰ型整合子对ARGs的污染传播扩散起到重要作用[10,11]。因此,本研究选择4种不同种类的ARGs、Ⅰ型整合子IntI1、16S rDNA 6个基因进行定性检测,4种ARGs包括四环素类ARGs-tetC、磺胺类ARGs-sulII、大环内酯类ARGs-ermB、β-内酰胺类ARGs-blaPSE-1。所用的引物序列、产物长度及PCR反应程序如表3所示。本实验所使用的引物均由生工生物工程(上海)股份有限公司合成。普通PCR实验体系采用25 μL反应体系:12.5 μL Premix Ex TaqTM Hot Start Version(Takara, 中国);各1 μL的上、下游引物;1 μL的DNA模板;9.5 μL的ddH2O。PCR产物使用质量浓度为1.5%的琼脂糖凝胶电泳(110 V,30 min)进行检测。普通PCR定性检测结果显示,YJ生活污水处理系统样本中存在tetC、sulII、ermB、blaPSE-1、IntI1、16S rDNA 6种基因,且琼脂糖凝胶电泳条带清晰明亮,如图2所示。
表3 PCR/qPCR引物序列及反应程序
Table 3 Primer sequences and reaction procedures of PCR/qPCR

目标基因引物序列(5′-3′)产物长度(bp)PCR/qPCR反应程序参考文献tetCF:TGCGTTGATGCAATTTCTATGC33595 ℃,1 min;(95 ℃,10 s;64 ℃,34 s;72 ℃,30 s)×40;[12]R:GGAATGGTGCATGCAAGGAG[72 ℃,10 min]∗sulIIF:TCCGGTGGAGGCCGGTATCTGG19195 ℃,2 min;(95 ℃,10 s;63 ℃,30 s;72 ℃,45 s)×40;[13]R:CGGGAATGCCATCTGCCTTGAG[72 ℃,10 min]∗ermBF:GATACCGTTTACGAAATTGG36495 ℃,3 min;(95 ℃,15 s;58 ℃,20 s;72 ℃,30 s)×40;[14]R:GAATCGAGACTTGAGTGTGC[72 ℃,10 min]∗blaPSE-1F:TGCTTCGCAACTATGACTAC43895 ℃,3 min;(94 ℃,30 s;55 ℃,30 s;72 ℃,60 s)×40;[15]R:AGCCTGTGTTTGAGCTAGAT[72 ℃,10 min]∗IntI1F:CCTCCCGCACGATGATC28094 ℃,9 min;(94 ℃,30 s;57.5 ℃,30 s;72 ℃,60 s)×35;本研究R:TCCACGCATCGTCAGGC[72 ℃,10 min]∗16S rDNAF:CCTACGGGAGGCAGCAG17895 ℃,3 min;(95 ℃,30 s;60 ℃,30 s;72 ℃,40 s)×40;[16]R:ATTACCGCGGCTGCTGG[72 ℃,10 min]∗
注:PCR/qPCR反应程序中[72 ℃,10 min]*为PCR的延伸过程,qPCR无需进行该过程。

图2 部分基因琼脂糖凝胶电泳结果(Marker:天根MarkerⅡ)
Fig.2 Results of agarose gel electrophoresis of some genes (Marker: Tian Gen MarkerⅡ)
1.5 实时荧光定量PCR定量检测
在普通PCR良好定性检出的情况下,考虑对6个基因进行实时荧光定量PCR试验。实时荧光定量PCR测定采用SYBR Green Ⅰ方法,试验过程在CFX-96定量PCR仪(Bio-Rad,美国)上完成。所使用的引物和升温程序如表3所示(qPCR反应程序无[72 ℃,10 min]*延伸过程)。定量PCR采用20 μL反应体系:10 μL SYBR Premix Ex TaqTM(Takara, 日本);1.0 μL DNA模板;各0.4 μL前、后引物;8.2 μL ddH2O。标准曲线采用10倍梯度稀释的已知浓度标准质粒为模板扩增获得,标准质粒采用对目标基因进行克隆的方法构建,具体过程参考Bai等[17]的研究。所有标准曲线线性拟合相关系数均在0.98以上,扩增效率在90%~110%为优。溶解曲线在一定温度段内得到单峰,表明标准质粒特异性良好,结果可靠。每个点位的污水胞内(胞外)DNA样品均设置2组平行,qPCR反应中每个样品均设置3个平行检测孔,因此,每个采样点样品的1种胞内(胞外)基因定量检测数据可得到6组。
1.6 数据统计分析
目前针对污水处理系统包括污水、污泥处理单元全流程的ARGs污染以及胞外ARGs分布、去除等方面的研究甚少,本文采用基因的绝对丰度、相对丰度、去除log值3个指标,衡量考察YJ生活污水处理系统污水、污泥处理工艺沿程各单元胞内、胞外目标基因的分布变化与去除效果,使用Excel 2016、Origin 2018 SR1等软件对试验测定数据进行分析,计算得到各样品胞内、胞外各基因的绝对丰度、相对丰度与去除log值。绝对丰度为单位体积污水样品或单位质量冻干污泥样品中所含有的目标基因的量,以copies/mL或copies/g干污泥计;相对丰度为同一DNA样品中目标基因的绝对丰度与16S rDNA的绝对丰度之比,无量纲;去除log值计算公式为lg(C0/Cx),其中:C0、Cx为某处理单元前、后污水的基因丰度,正值表示去除减少,负值表示富集增加,无量纲。相关性分析则利用Past 3.0软件完成,分析采用Pearson相关,相关性程度用P值来衡量,若P<0.01,则认为显著性相关程度极高;若0.01<P<0.05,则认为显著性相关程度较高;若P>0.05,则认为无显著相关性。
2 结果与讨论
2.1 污水处理系统中胞内、胞外基因绝对丰度分布及去除log值
YJ生活污水处理系统污水、污泥处理工艺沿程各单元胞内和胞外ARGs、Ⅰ型整合子IntI1、16S rDNA绝对丰度如图3所示。除脱水前、后污泥的绝对丰度单位为copies/g干污泥,其余均为copies/mL。下文选择重点单元进行分析探讨。

![]() tetC;
tetC; ![]() sulII
sulII![]() ermB;
ermB; ![]() blaPSE-1;
blaPSE-1; ![]() Intl1;
Intl1; ![]() 16S rDNA。
16S rDNA。
图3 YJ污水处理系统胞内、胞外基因绝对丰度
Fig.3 Absolute abundance of intracellular, extracellular genes in YJ wastewater treatment plant
2.1.1 污水处理工艺
由图3可知:在总进水中,胞内ARGs的绝对丰度占有绝对主导地位,4种ARGs绝对丰度的数量级为106~107(胞内)、100~102(胞外),胞内ARGs总绝对丰度达到1.30×108 copies/mL,胞外ARGs总绝对丰度则为2.09×102 copies/mL。
在经过细格栅与旋流沉砂池作用后,污水胞内tetC、sulII、ermB、blaPSE-1、IntI1绝对丰度较总进水分别下降了0.32log、-0.28log、0.24log、0.17log、0.08log,总胞内ARGs绝对丰度去除0.09log,张明美[18]对多个生活污水系统的研究发现,经过沉砂池后,污水中ARGs(胞内)的削减量很小,与本研究结果相同,但其并未研究胞外ARGs的分布变化。胞外tetC、sulII、ermB、IntI1绝对丰度较总进水分别上升0.26log、0.15log、1.44log、0.59log,胞外ermB大幅度上升,胞外blaPSE-1在此单元出水得以检出,绝对丰度为6.12×102 copies/mL。总胞外ARGs绝对丰度上升0.69log。胞外16S rDNA在此单元大量富集,可能由于泥区废液总回流液混入了进水,导致其绝对丰度上升2.87log。可以发现,该单元对胞内基因具有较小的去除作用,而对胞外基因具有明显的富集作用。
污水经过A2/O生化池作用后,tetC、sulII、ermB、blaPSE-1、IntI1胞内绝对丰度较旋流沉砂池出水分别下降了1.01log、0.37log、0.91log、0.89log、0.63log。胞外基因变化趋势因基因而异,经过A2/O生化池作用后,tetC、sulII、IntI1胞外绝对丰度分别较旋流沉砂池出水上升了0.71log、1.27log、1.17log,而胞外ermB、blaPSE-1、16S rDNA则下降了0.53log、0.91log、0.64log。在生化池中,胞外基因可能与生化池微生物发生了强烈的水平基因转移,而微生物对不同的胞外基因作用效果不同,猜测可能胞外ermB、blaPSE-1、16S rDNA更易被微生物获取发生“转化”而下降,而胞外tetC、sulII、IntI1可能是由于微生物细胞释放胞内基因而增加。
经过二沉池后,各胞内基因都随着污泥沉降而大幅减少,胞内tetC、sulII、ermB、blaPSE-1、IntI1绝对丰度较生化池出水去除log值分别达到0.90log、1.06log、1.33log、0.47log、0.88log,16S rDNA下降了2个数量级,总胞内ARGs绝对丰度较生化池出水去除log值达到1.05log,二沉池出水总胞内ARGs绝对丰度为2.50×106 copies/mL。张明美[18]同样发现,二沉池的污泥沉降作用可以大幅度减少ARGs的总量。而胞外基因则与胞内基因完全相反,均发生了负去除现象,胞外tetC、sulII、ermB、blaPSE-1、IntI1绝对丰度较生化池出水分别增长了0.77log、0.64log、1.09log、0.63log、1.43log。胞外16S rDNA绝对丰度也上升了1.72log,可能是二沉池中泥龄较老的微生物细胞发生破壁现象,导致微生物细胞内DNA释放至胞外,从而导致二沉池出水中的胞外基因全面升高。二沉池对ARGs的去除作用应同时结合胞内、胞外ARGs的前后变化来评判,张明美[18]的研究仍具有一定的局限性。
投加PAM、PAC进行絮凝沉淀后,微生物量持续下降,出水胞内基因再次明显下降,而絮凝沉淀作用对胞外基因作用并不十分明显。
对于ClO2消毒出水,胞内tetC、ermB绝对丰度较上一单元出水产生轻微富集作用,胞内sulII、blaPSE-1、IntI1、16S rDNA均下降了0.29log、0.20log、0.60log、0.26log。最终出水胞内tetC、sulII、ermB、blaPSE-1、IntI1、16S rDNA相较于总进水的去除log值为2.23log、1.70log、2.77log、2.64log、2.09log、2.32log,总胞内ARGs去除log值为2.10log,绝对丰度达到4.13×105,5.58×105,5.30×104,2.46×103,6.70×105,2.03×107 copies/mL。有研究表明,紫外/氯化工艺或UV/H2O2工艺等消毒技术可对出水中抗生素抗性菌ARBs产生较好的削减杀灭作用[19,20],但此研究忽视了消毒技术可能会导致抗性菌ARBs细胞破裂而释放出胞外ARGs的现象。有报道称在太湖地表水体中对tetA、tetC进行检测,其胞内绝对丰度高达104~105 copies/mL[21],可见本研究的污水处理系统出水中胞内tetC仍可能会增大地表水流域的抗性污染。本课题组有前续研究结果显示,杭州湾区域1座氧化沟为主体的污水处理系统(20%生活污水+80%工业废水)对胞内ermB、blaPSE-1、IntI1、16S rDNA的去除log值达到2.04log、1.38log、1.38log、1.64log[22],各基因去除率均低于该生活污水处理系统,可能由于工业废水的存在,微生物群落比单纯生活污水的微生物群落更复杂而更难去除,对于胞外基因的分布、去除仍未关注。奚慧[23]在上海黄浦江上游水体中对sulII进行检测,胞内绝对丰度高达3.43×106 copies/mL,该污水处理系统出水低于此一个数量级,表明去除效果良好。吴韵斐等[24]在上海市金泽水源型水库中检出磺胺类ARGs绝对丰度为3.86×105~4.23×106 copies/mL,亦证明该污水处理系统出水安全性尚可。消毒操作对胞外基因产生了富集作用,胞外tetC、sulII、ermB、blaPSE-1、16S rDNA绝对丰度较上一单元出水上升了1.16log、0.56log、0.66log、0.35log、0.16log,可能消毒氧化使微生物细胞破壁释放出胞内DNA。消毒对胞外IntI1作用不大,减少了0.08log。最终出水胞外tetC、sulII、ermB、IntI1、16S rDNA相较于总进水增加了3.30log、3.19log、2.95log、3.10log、3.79log,胞外blaPSE-1相较于旋流沉砂池出水去除了0.12log,总胞外ARGs增加了3.20log值,胞外tetC、sulII、ermB、blaPSE-1、IntI1、16S rDNA绝对丰度达到4.11×104,2.88×105,3.97×103,4.66×102,6.73×104,2.50×106 copies/mL,总胞外ARGs绝对丰度仅比总胞内ARGs绝对丰度低0.49个数量级。
该生活污水处理系统对胞内ARGs绝对丰度整体表现为持续下降的趋势,表明该污水处理流程对胞内ARGs具有一定的削减去除作用,能够降低进水的抗性风险。与本文研究结果相似,已有较多研究报道的生活污水处理系统表现出较好的胞内ARGs去除能力,我国北方2座以A2/O为主体工艺的污水处理系统对胞内ARGs去除率高达89.9%~99.8%[25],1座三沟式氧化沟污水处理系统和1座A2/O对胞内ARGs的去除log值分别可达到1.10log~2.25log、1.16log~2.44log[26]。但对污水处理系统污水处理流程胞外ARGs的分布变化研究甚少,该污水处理系统会使出水胞外ARGs绝对丰度有极大的提高,出水中大量的胞外ARGs进入生态环境,可能会与环境中的微生物发生水平基因转移,导致抗生素抗性的传播扩散,具体的传播机制有待进一步研究。
2.1.2 污泥处理工艺
由图3可知:泥区废液总回流液、二沉池污泥回流液、脱水机房冲洗液(含/不含污泥脱水滤液)、污泥浓缩池上清液胞内ARGs绝对丰度达到106~108数量级。目前泥区废液总回流液的回流操作、污泥脱水压滤操作对污水处理系统ARGs的影响仍研究较少。本研究中,泥区废液总回流液的胞内ARGs绝对丰度低于总进水,回流有利于进行胞内ARGs的稀释。但对于胞外ARGs绝对丰度,污泥处理流程单元总胞外ARGs绝对丰度可达104~105数量级,远大于总进水,回流操作会增大总进水的胞外ARGs负荷,这就解释了旋流沉砂池出水胞外ARGs绝对丰度远大于总进水的原因。对于脱水前、后污泥,各基因及总胞内ARGs绝对丰度基本保持不变,可以判断污泥脱水压滤机基本没有使细胞发生破壁现象,胞内基因并没有释放至胞外。
2.2 污水处理系统中胞内、胞外基因相对丰度分布及去除log值
YJ生活污水处理系统污水处理工艺、污泥处理工艺沿程各单元胞内和胞外ARGs、Ⅰ型整合子IntI1的相对丰度如图4所示。下文选择重点单元进行分析探讨。
![]() tetC;
tetC;![]() suIII;
suIII; ![]() ermB;
ermB; ![]() blaPSE-1;
blaPSE-1; ![]() IntI1。
IntI1。
图4 YJ污水处理系统胞内、胞外基因相对丰度
Fig.4 Relative abundance of intracellular, extracellular genes in YJ wastewater treatment plant
2.2.1 污水处理工艺
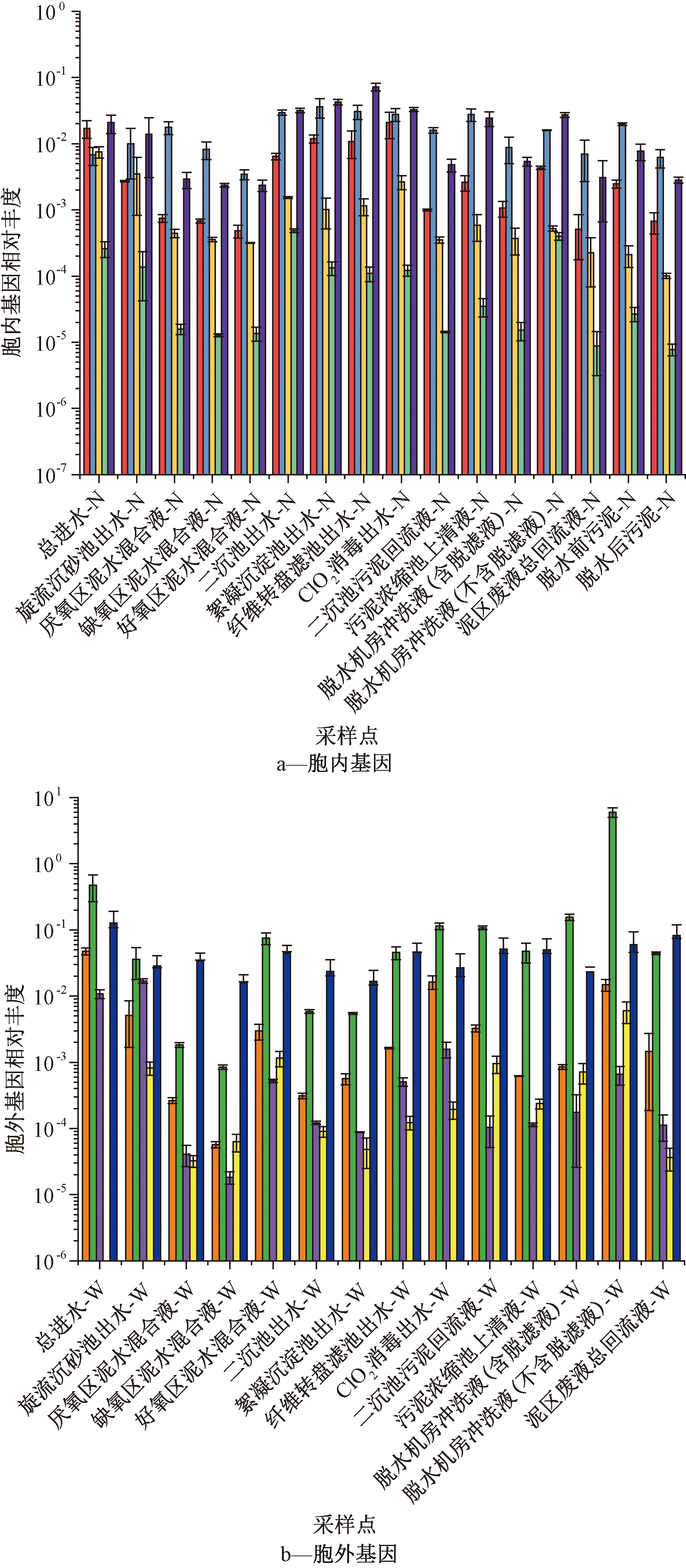
由图4可知:在污水处理系统污水处理流程的总进水中,胞外ARGs的相对丰度远高于胞内ARGs相对丰度,4种ARGs相对丰度的数量级为10-4~10-2(胞内)、10-2~10-1(胞外),胞内ARGs总相对丰度达到3.16×10-2,胞外ARGs总相对丰度则为5.29×10-1。通过高相对丰度的胞外ARGs可以判断,自然环境中存在大量携带有抗生素抗性基因的DNA片段,易导致水平基因转移的发生及抗性风险的传播。
在经过细格栅与旋流沉砂池作用后,污水胞内tetC、sulII、ermB、blaPSE-1、IntI1相对丰度较总进水分别下降了0.80log、-0.17log、0.33log、0.28log、0.17log,胞外tetC、sulII、ermB、IntI1相对丰度较总进水分别下降了0.98log、1.12log、-0.20log、0.64log。胞外blaPSE-1在此单元出水得以检出,相对丰度为8.20×10-4。该单元作用对胞外基因相对丰度的去除作用强于胞内基因。
污水经过A2/O生化池作用后,tetC、sulII、ermB、blaPSE-1、IntI1胞内相对丰度较旋流沉砂池出水分别下降了0.75log、0.46log、1.04log、1.00log、0.77log。胞外基因变化趋势因基因而异,经过A2/O生化池作用后,胞外ermB的相对丰度大幅削减,较旋流沉砂池出水下降了1.51log,胞外tetC的相对丰度下降了0.23log,其余基因均有所上升,sulII、blaPSE-1、IntI1均上升了0.32log、0.15log、0.19log。生化池能够高效降低大环内酯类抗性基因ermB的风险水平。张衍等[5]对1座采用A/O工艺的生活污水处理系统进行研究发现,ARGs的单位质量DNA拷贝数浓度较进水下降了1个数量级,这一结果与本研究对tetC、ermB、blaPSE-1在A2/O工艺中的研究结果类似,说明生化单元可以降低部分ARBs在微生物群落中比例。
经过二沉池后,与绝对丰度的情况截然相反,胞内tetC、sulII、ermB、blaPSE-1、IntI1相对丰度较生化池出水上升了1.13log、0.93log、0.68log、1.55log、1.13log,总胞内ARGs相对丰度较生化池出水上升了0.95log,二沉池出水总胞内ARGs相对丰度为3.81×10-2。而胞外tetC、sulII、ermB、blaPSE-1、IntI1相对丰度较生化池出水分别降低了0.98log、1.11log、0.63log、1.11log、0.28log。总胞外ARGs相对丰度较生化池出水降低了1.10log,二沉池出水总胞外ARGs相对丰度为6.40×10-3。综合绝对丰度与相对丰度的结果,在进入下一单元的水体中,胞内微生物量少却含有较高比例的ARGs,胞外DNA量大却含有较低比例的ARGs,二者均具有较高风险。
投加PAM、PAC进行絮凝沉淀后,对胞内blaPSE-1相对丰度作用较为明显,较上一单元出水下降了0.56log,tetC、sulII、IntI1有所增加,ermB有所降低。对胞外tetC较上一单元出水增加了0.26log,对胞外blaPSE-1相对丰度下降了0.27log,其余胞外基因略有下降。
纤维转盘滤池单元对胞内基因相对丰度作用不明显,而对胞外基因相对丰度富集作用较强,胞外tetC、sulII、ermB、blaPSE-1、IntI1相对丰度较絮凝沉淀池出水分别增加了0.46log、0.92log、0.76log、0.41log、0.44log。总胞外ARGs相对丰度较上一级出水增加了0.89log。
ClO2消毒出水对各基因的相对丰度影响不一,可能与基因种类有关。已有研究表明,消毒剂量是影响ARGs削减效果的重要因素,CT值(消毒剂浓度×接触时间)的变化对不同ARGs的削减效果影响不同,甚至可能会产生负作用[27]。胞内tetC、sulII、ermB、blaPSE-1、IntI1相对丰度较纤维转盘滤池出水去除log值达到-0.29log、0.04log、-0.36log、-0.05log、0.34log,最终出水胞内tetC、sulII、ermB、blaPSE-1、IntI1相较总进水的去除log值为-0.09log、-0.61log、0.45log、0.33log、-0.20log,各基因相对丰度达到2.09×10-2、2.78×10-2、2.65×10-3、1.22×10-4、3.92×10-2,总胞内ARGs相对丰度为5.14×10-2,高于总进水。Ling等[28]在中国南方北江河中检测胞内sulII相对丰度均值为1.58×10-3,若将其作为环境水体本底值,则该污水处理系统外排水胞内sulII基因丰度高于环境水体,会引起环境风险增加。同样,杨继平等[29]在天津海河地表水体中检出tetC相对丰度为1.4×10-4~1.1×10-3,低于该污水处理系统出水胞内tetC相对丰度1个数量级。胞外tetC、sulII、ermB、blaPSE-1、IntI1相对丰度较纤维转盘滤池出水去除log值达到-1.00log、-0.40log、-0.49log、-0.20log、0.24log,最终出水胞外tetC、sulII、ermB、IntI1相较于总进水的去除log值为0.47log、0.61log、0.84log、0.67log,胞外blaPSE-1较旋流沉砂池出水下降了0.63log。消毒出水tetC、sulII、ermB、blaPSE-1、IntI1相对丰度分别达到1.64×10-2、1.15×10-1、1.58×10-3、1.94×10-4、2.68×10-2,总胞外ARGs相对丰度为1.33×10-1,低于总进水。
在该生活污水处理系统综合作用后,出水总胞内ARGs相对丰度不降反升,虽然该系统能够使胞内ARGs绝对丰度显著降低,但出水中含有的具有耐药性的菌株比例增加,即污水厂作为1种选择压力,使具有高抗性的耐药微生物进入生态环境,李金梅等[30]也得出关于胞内ARGs相对丰度变化的相似结论。而对于胞外ARGs相对丰度,该污水处理系统能够降低其水平,但是由于存在的胞外ARGs绝对丰度过大,在环境中的胞外ARGs综合风险水平亦较高。有研究表明,现有污水处理工艺对耐药菌株和ARGs有一定的削减作用,有助于降低ARGs的传播风险,但是残留于出水中的耐药菌株和ARGs丰度依然较高[31]。
2.2.2 污泥处理工艺
泥区废液总回流液、二沉池污泥回流液、脱水机房冲洗液(含/不含污泥脱水滤液)、污泥浓缩池上清液胞内、总胞外ARGs相对丰度达到10-3~10-2数量级、10-2~100数量级。泥区废液总回流液的胞内、胞外ARGs相对丰度均低于总进水,回流有利于进行胞内、胞外ARGs相对丰度的稀释。
2.3 基因、水质因子的相关性
将污水、污泥处理工艺沿程各单元的tetC、sulII、ermB、blaPSE-1、IntI1、16S rDNA的胞内外绝对丰度、相对丰度和表2所示的样品水质因子pH、COD、NH3-N等数据进行相关性分析,主要结果如下。
对污水处理工艺沿程各单元胞内基因绝对丰度、水质因子相关性分析发现,tetC(P<0.01,r=0.9824)、ermB(P<0.01,r=0.9889)、blaPSE-1(P<0.01,r=0.9815)均与IntI1呈显著正相关,且这3个ARGs彼此之间也呈显著相关性,可以判断IntI1基因在污水处理工艺沿程的tetC、ermB、blaPSE-1的传播扩散中起到了较强作用,且这3种ARGs的传播机制可能类似。sulII(P<0.05,r=0.7620)、IntI1(P<0.05,r=0.7287)均与16S rDNA呈显著正相关,表明sulII与IntI1在污水处理工艺中的变化与微生物总量的变化有关。16S rDNA(P<0.01,r=-0.8947)与污水pH呈显著负相关,表明污水的pH值升高,其微生物总量会出现下降现象。tetC(P<0.01,r=0.9668)、ermB(P<0.01,r=0.9735)、blaPSE-1(P<0.01,r=0.9750)、IntI1(P<0.01,r=0.9805)与污水COD呈显著正相关,表明这4种基因受污水中有机物浓度影响,随着有机物被逐步去除,基因浓度也随之下降。类似地,除了sulII基因,tetC(P<0.01,r=0.8990)、ermB(P<0.01,r=0.9141)、blaPSE-1(P<0.01,r=0.8946)、IntI1(P<0.01,r=0.9595)、16S rDNA(P<0.01,r=0.8360)均与污水中NH3-N呈显著正相关,进一步说明sulII的传播机制与其余3个ARGs不同。有研究指出,sulII基因通常存在于质粒上,不会被IntI1基因所捕获[32],与此分析结果相吻合。
对污水处理工艺沿程各单元胞外基因绝对丰度、水质因子的相关性分析发现,tetC(P<0.01,r=0.9880)、sulII(P<0.01,r=0.9967)均与ermB呈显著正相关,说明这3个基因在细胞外的传播机制可能类似。IntI1与污水的COD(P<0.01,r=-0.8200)呈显著负相关,同时与污水的NH3-N(P<0.05,r=-0.7255)呈显著负相关,16S rDNA也与COD(P<0.05,r=-0.7210)、NH3-N(P<0.05,r=-0.6793)呈显著负相关,表明污水在被逐级处理的同时,水体中的胞外DNA逐渐增多,可能胞内DNA由于细胞破壁释放至胞外所致。
对污泥处理工艺沿程各单元胞内基因丰度、水质因子相关性分析发现:blaPSE-1绝对丰度(P<0.01,r=0.9631)和blaPSE-1相对丰度(P<0.05,r=0.8848)与NH3-N呈显著正相关,说明去除NH3-N,可以同时降低胞内blaPSE-1的绝对丰度和相对丰度。
对污泥处理工艺沿程各单元胞外基因相对丰度、水质因子相关性分析发现,tetC(P<0.01,r=0.9571)、sulII(P<0.01,r=0.9080)、ermB(P<0.01,r=0.8851)、blaPSE-1(P<0.01,r=0.9234)均与NH3-N呈显著正相关,也表明水体中NH3-N减少可能降低污泥处理流程中胞外ARGs在胞外DNA中的占比,从而降低风险。
3 结 论
1)本研究发现在河北省某A2/O生活污水处理系统存在抗性风险。在污水处理系统中对抗生素抗性基因tetC、sulII、ermB、blaPSE-1,Ⅰ型整合子IntI1,16S rDNA进行了检测。总进水的总ARGs绝对丰度达到1.30×108 copies/mL(胞内)、2.09×102 copies/mL(胞外),总ARGs相对丰度达到3.16×10-2(胞内)、5.29×10-1(胞外)。ARGs作为一种新型污染物“汇”入生活污水处理系统。
2)污水处理最终出水的总ARGs绝对丰度去除log 值达到2.10log(胞内去除)、-3.20log(胞外富集),总ARGs相对丰度去除log 值达到-0.21log(胞内富集)、0.60log(胞外去除)。该污水处理系统作为ARGs的“源”进入生态环境,可能会威胁人类健康与生态安全。靶向设计同时去除水体胞内、胞外ARGs 绝对丰度、相对丰度的污水处理技术是减少乃至消灭抗性污染的关键,有待进一步研究。
3)泥区废液总回流液的回流有助于总进水胞内ARGs绝对丰度、相对丰度和胞外ARGs相对丰度的降低,但会造成胞外ARGs绝对丰度增加,增大总进水的胞外ARGs负荷。
4)相关性研究表明,Ⅰ型整合子IntI1可能会促进tetC、ermB、blaPSE-1在污水处理系统中的传播,控制污水水质可能会影响ARGs的分布与扩散。
[1] Brandt K K, Amézquita A, Backhaus T, et al.Ecotoxicological assessment of antibiotics: a call for improved consideration of microorganisms[J].Environment International, 2015, 85: 189-205.
[2] 董祎, 张万筠, 鲍晨宁, 等.抗生素抗性基因在环境中的来源与传播[J].辽宁化工,2018,47(3): 253-255.
[3] 苏建强, 黄福义, 朱永官.环境抗生素抗性基因研究进展[J].生物多样性, 2013, 21(4): 481-487.
[4] Qiao M, Ying G G, Singer A C, et al.Review of antibiotic resistance in China and its environment[J].Environment International, 2018, 110: 160-172.
[5] 张衍, 陈吕军, 谢辉, 等.两座污水处理系统中细胞态和游离态抗生素抗性基因的丰度特征[J].环境科学, 2017,38(9): 273-280.
[6] Andersson D I, Hughes D.Antibiotic resistance and its cost: is it possible to reverse resistance? [J].Nature Reviews Microbiology, 2010, 8(4): 260-271.
[7] Czekalski N, Diez E G, Burgmann H.Wastewater as a point source of antibiotic-resistance genes in the sediment of a freshwater lake [J].ISME Journal, 2014, 8 (7): 1381-1390.
[8] Rizzo L, Manaia C, Merlin C, et al.Urban wastewater treatment plants as hotspots for antibiotic resistant bacteria and genes spread into the environment: a review [J].Science of the Total Environment, 2013, 447: 345-360.
[9] Eichmiller J J, Best S E, Sorensen P W.Effects of temperature and trophic state on degradation of environmental DNA in lake water [J].Environmental Science and Technology, 2016, 50(4), 1859-1867.
[10] 苏志国, 张衍, 代天娇, 等.环境中抗生素抗性基因与Ⅰ型整合子的研究进展[J].微生物学通报, 2018, 45(10): 153-169.
[11] Ma L P, Li A D, Yin X L, et al.The prevalence of integrons as the carrier of antibiotic resistance genes in natural and man-made environments[J].Environmental Science and Technology, 2017,51:5721-5728.
[12] Chen B W, Liang X M, Huang X P, et al.Differentiating anthropogenic impacts on ARGs in the Pearl River Estuary by using suitable gene indicators[J].Water Research, 2013, 47(8): 2811-2820.
[13] Jiang L, Hu X L, Xu T, et al.Prevalence of antibiotic resistance genes and their relationship with antibiotics in the Huangpu River and the drinking waters sources, Shanghai, China[J].Science of the Total Environment, 2013, 458-460: 267-272.
[14] Huerta B, Marti E, Gros M, et al.Exploring the links between antibiotic occurrence, antibiotic resistance, and bacterial communities in water supply reservoirs[J].Science of the Total Environment, 2013, 456/457(7): 161-170.
[15] Stoll C, Sidhu J P S, Tiehm A, et al.Prevalence of clinically relevant antibiotic resistance genes in surface water samples collected from Germany and Australia [J].Environmental Science and Technology, 2012, 46(17): 9716-9726.
[16] He S, Gall D L, Mcmahon K D.Candidatus Accumulibacter" population structure in enhanced biological phosphorus removal sludges as revealed by polyphosphate kinase genes[J].Applied and Environmental Microbiology, 2007, 73(18): 5865-5874.
[17] Bai Y H, Sun Q H, Wen D H, et al.Abundance of ammonia-oxidizing bacteria and archaea in industrial and domestic wastewater treatment plants [J].FEMS Microbiology Ecology, 2012, 80(2): 323-330.
[18] 张明美.污水处理系统中抗生素抗性基因污染研究[D].杭州:浙江大学,2013.
[19] Zhang Y Y, Zhuang Y, Geng J J, et al.Inactivation of antibiotic resistance genes in municipal wastewater effluent by chlorination and sequential UV/chlorination disinfection [J].Science of the Total Environment, 2015, 512: 125-132.
[20] Ferro G, Guarino F, Castiglione S, et al.Antibiotic resistance spread potential in urban wastewater effluents disinfected by UV/H2O2 process [J].Science of the Total Environment, 2016, 560: 29-35.
[21] Zhang X X, Wu B, Zhang Y, et al.Class 1 integronase gene and tetracycline resistance genes tetA and tetC in different water environments of Jiangsu Province, China[J].Ecotoxicology, 2009, 18(6): 652-660.
[22] 李奥林.污水处理系统和受污染水环境中抗生素抗性基因污染研究[D].北京:清华大学, 2018.
[23] 奚慧.上海城市典型水体中抗生素抗性基因时空分布特征及其影响因子研究[D].上海:华东师范大学, 2018.
[24] 吴韵斐,何义亮,袁其懿,等.水源型水库抗生素抗性基因赋存特征研究[J].环境科学学报,39(6):1834-1841.
[25] Mao D Q, Yu S, Rysz M, et al.Prevalence and proliferation of antibiotic resistance genes in two municipal wastewater treatment plants [J].Water Research, 2015, 85: 458-466.
[26] Li J N, Cheng W X, Xu L K, et al.Occurrence and removal of antibiotics and the corresponding resistance genes in wastewater treatment plants: effluents’influence to downstream water environment [J].Environmental Science and Pollution Research, 2016, 23(7): 6826-6835.
[27] 翟文超, 罗义, 赵静, 等.抗生素抗性基因在典型行业和市政污水中的污染特征及消减研究进展[J].环境化学,2014,33(2): 206-216.
[28] Ling Z H, Yang Y, Huang Y L, et al.A preliminary investigation on the occurrence and distribution of antibiotic resistance genes in the Beijiang River, South China[J].Journal of Environmental Sciences, 2013, 25(8): 1656-1661.
[29] 杨继平, 邱志刚, 袁兆康,等.天津海河流域抗生素抗性基因分布特征及与指示微生物的关系[J].环境与健康杂志, 2017, 34(4): 313-316,376.
[30] 李金梅,李曦,张舒婷.消毒工艺对水体中抗生素抗性基因的去除效果[J].净水技术, 2018,37(2): 10-16.
[31] Gao P, Munir M, Xagoraraki I.Correlation of tetracycline and sulfonamide antibiotics with corresponding resistance genes and resistant bacteria in a conventional municipal wastewater treatment plant[J].Science of the Total Environment,2012,421/422(3): 173-183.
[32] Chen B W, Liang X M, Nie X P, et al.The role of class I integrons in the dissemination of sulfonamide resistance genes in the Pearl River and Pearl River Estuary, South China[J].Journal of Hazardous Materials, 2015, 282: 61-67.