0 引 言
畜禽养殖业中抗生素高浓度残留现象已成为国内外社会关注的热点问题[1,2]。近年来,国内外研究发现与物化处理工艺和活性污泥技术相比,人工湿地对养殖污水中抗生素展现出较高和更稳定的处理效果,并具有较低处理成本的优势[3,4]。人工湿地对污水中抗生素的高效去除得益于该工艺所具有的多途径污染物降解转化特征,其可通过植物吸收、填料吸附和微生物降解等作用去除污水中抗生素成分,同时该工艺较长的水力停留时间还可强化抗生素水解降解效果[3,5]。然而,现有文献报道尚未清楚定量上述不同去除途径在抗生素削减过程中起到的作用程度,因此本文针对此问题,初步分析探讨了水解作用、吸附作用、生物作用(包括植物作用和微生物作用)在抗生素去除过程中各自的贡献率,以期为进一步理解抗生素在人工湿地中行为特征提供参考。
1 材料与方法
1.1 实验设计
本论文设计不同试验装置构型与条件,通过检测不同系统对抗生素的去除效果,基于削减差量比较方法确定水解作用、吸附作用、生物作用(包括植物作用和微生物作用)在抗生素去除过程中各自的贡献率。为实现不同作用的差异性比较,本文设置了4套实验装置(图1中A/B/C/D),具体试验条件设计如下:A装置不添加任何植物和填料,以考察水解作用能力;B装置添加紫外灭菌后的湿地填料,不栽种植物,以考察水解作用和吸附作用的协同效果;C装置添加未灭菌湿地填料,并通过对比B装置,以考察微生物作用的贡献程度;D装置则添加植物和未灭菌湿地填料,考察上述多种作用的协同处理效率。4套实验装置皆以序批式条件运行,并基于抗生素去除效率检测结果,根据差异度比较计算各去除途径在抗生素削减过程中的作用程度。同时,为确定畜禽养殖污水中传统污染物(有机物和氮磷营养盐)对抗生素去除过程的影响,依据上述实验设计分为2个处理组进行。处理组1所用模拟污水中仅含抗生素,未添加有机物和营养盐;处理组2所用模拟污水为抗生素、有机物和营养盐混合配制而成。处理组1中4套实验系统编号分别设定为R1、R2、R3和R4,而处理组2中4套实验系统编号分别设定为RM1、RM2、RM3和RM4。
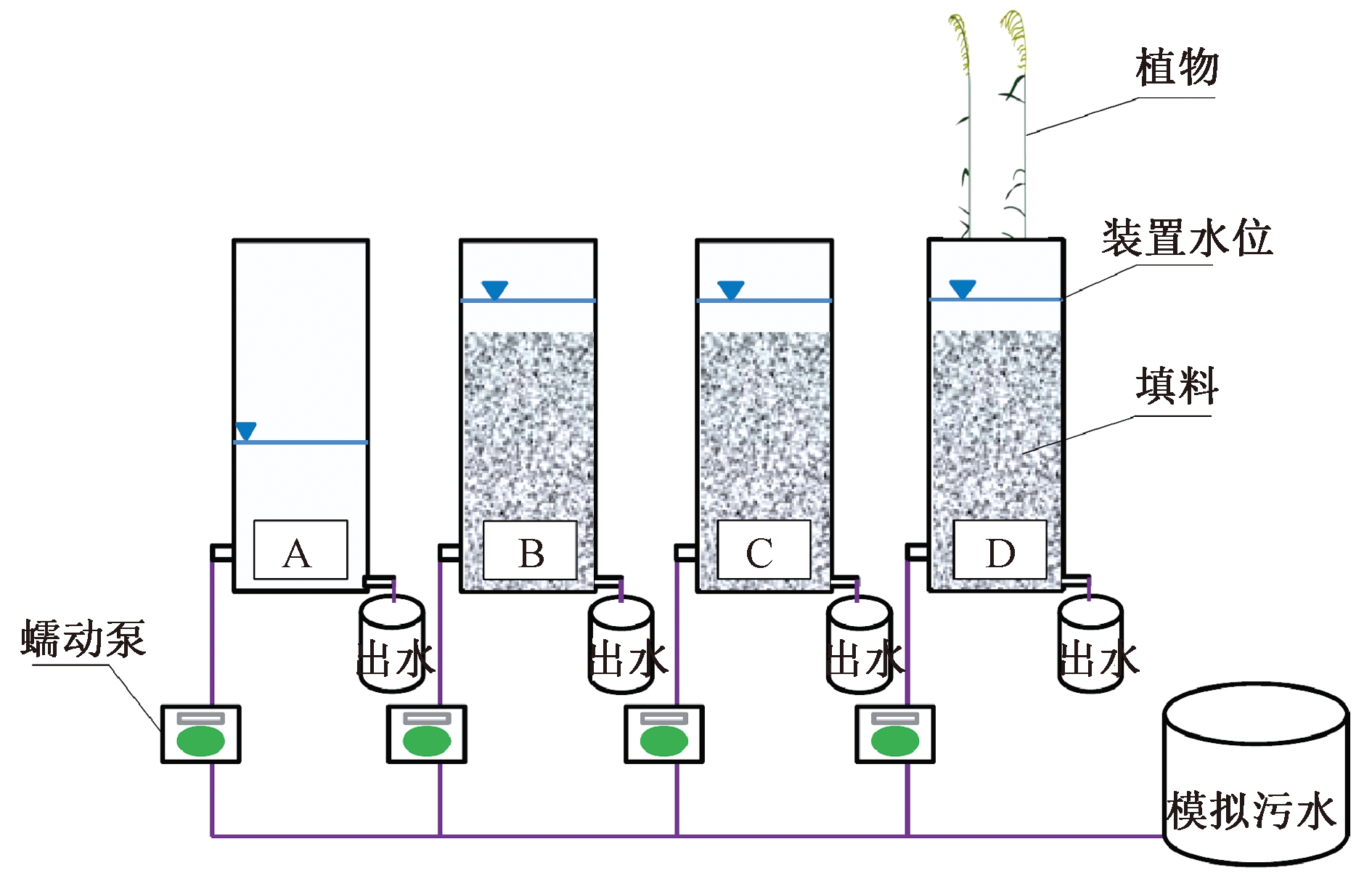
图1 实验所用装置示意
Figure 1 Schematic diagram of the designed experimental reactor
1.2 实验条件与材料
实验装置为有机玻璃圆柱体,柱体装置高为20 cm,直径为8.8 cm,有效容积约为1.2 L。排水口设置在装置底部,进水口设置在装置侧面,装置顶部安装有机玻璃盖体,并通过法兰固定封闭。实验装置上部盖体部分设置通气口和植物装备口,通气口以无机滤膜(0.45 μm)连接,降低气体交换过程中外界微生物的干扰作用,植物装备口在植物移栽后用封口膜封闭植物与其之间的缝隙。实验装置为序批式方法运行,每组实验运行4个周期,每周期时间设定为7 d,各装置单周期入水量统一设定为0.5 L,实验环境温度为(20±2) ℃。实验所用湿地填料(海蛎壳)取自研究组的水平潜流中试平台,湿地填料依据实验设计方案,经破碎筛分及部分灭菌处理后分别添加到3套实验系统(B/C/D)内部。实验所用湿地植物为芦苇。前期文献报道指出四环素类、氟喹诺酮类和磺胺类抗生素为畜禽养殖污染中具有较高残留浓度和较高检出频率的典型兽用抗生素[6],因此本文选用土霉素(OTC)、环丙沙星(CIP)和磺胺二甲基嘧啶(SMZ)作为目标抗生素,3种抗生素在模拟污水中添加浓度均设定为30 μg/L。
1.3 分析项目和方法
实验过程中抗生素浓度测定方法如下:定量采集各系统出水,加入0.05 g乙二胺四乙酸二钠,用6 mol/L的盐酸调节水样pH至3.0,并用0.45 μm孔径滤膜过滤;之后取定量过滤后水样与无水甲醇以体积比8∶2混合,用于后续分析;抗生素含量利用高效液相色谱-三重四级杆线性离子阱质谱仪(ABI 3200Q TRAP)进行测定,目标抗生素的测试参数参考文献[7]设定。实验装置出水量采用体积测量法测定。由于实验过程中各系统存在水量损失,因此对不同系统抗生素的去除效率评定依据水量平衡计算。
2 实验结果
2.1 抗生素在模拟系统中的去除效果
目标抗生素在2个处理组出水中皆有检出,其在不同实验设置条件下的出水浓度情况如图2所示。实验结果表明:A实验装置出水中3种抗生素含量皆高于B、C和D装置,OTC、CIP和SMZ在A实验装置出水中的浓度分别为20.41~25.48,25.90~28.19,27.36~28.77 μg/L,而在其他3套实验装置出水中的浓度则为0.89~17.33 μg/L。单一处理条件下,B、C和D实验装置出水中抗生素浓度较为相似,只有RM2系统出水中SMZ浓度(14.34~15.84 μg/L)显著高于RM3和RM4系统(9.95~12.82 μg/L和9.71~13.72 μg/L)。抗生素出水浓度会受到污水中传统污染物的影响,其中A装置出水中OTC浓度、C和D装置出水中SMZ浓度受此影响较为明显。R1和RM1系统出水中OTC浓度分别为20.41~23.48 μg/L和23.63~25.48 μg/L,而SMZ在R3、R4、RM3和RM4系统出水中的浓度分别为16.28~17.15,15.88~16.48,9.95~12.82,9.71~13.72 μg/L。对比3种典型目标抗生素,R1~R4系统的出水OTC浓度皆为最低(0.89~23.48 μg/L),处理组2的RM1~RM4系统出水中OTC浓度也存在类似规律(3.29~25.48 μg/L),而SMZ在处理组1和处理组2的B、C和D装置出水中浓度皆为最高(9.71~17.53 μg/L)。

图2 实验周期内不同系统出水中OTC、CIP和SMZ的浓度情况
Figure 2 The concentrations of OTC, CIP and SMZ in the effluent from different systems in running periods
2.2 湿地抗生素去除途径的作用程度
根据上述抗生素去除检测结果,对湿地主要抗生素去除途径进行了相对定量分析。由于OTC和CIP在C和D实验装置内去除差异度较小,因此本实验无法准确区分植物和微生物对两者去除的贡献率,故以生物作用形式来综合表征植物和微生物在OTC和CIP去除过程中的作用程度,而对于SMZ仍以4种作用进行分析(图3)。结果表明,水解和填料吸附在抗生素去除过程中的作用程度最为稳定,两者在4个周期内的各个实验系统内皆有显著的贡献表征。生物作用在OTC和CIP去除过程中作用较弱,而其所包括的植物作用和微生物作用分别为处理组1和处理组2中SMZ的主要去除途径。为更加直观地体现不同去除途径对3种抗生素的作用效果,将抗生素去除途径的相对平均贡献比例总结见表1。
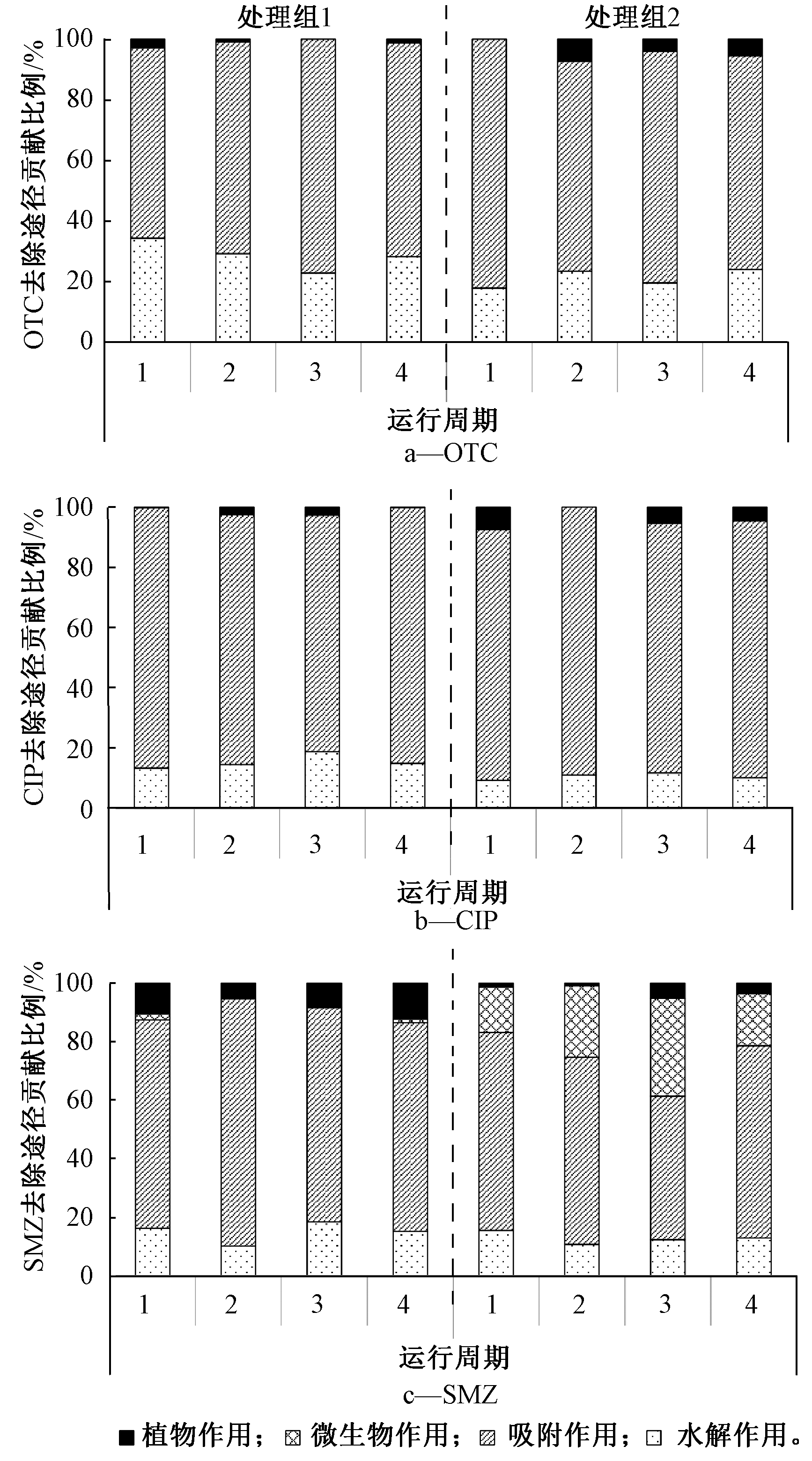
图3 人工湿地中抗生素不同去除途径的相对作用程度
Figure 3 The performance of antibiotic different removal pathways in constructed wetland
途径分析结果表明,OTC和CIP去除途径较为相似,填料吸附作用为其主要去除途径,对于OTC和CIP去除的平均贡献比例>70%。传统污染物对OTC和CIP吸附过程影响也较为一致,传统污染物存在导致吸附作用对OTC和CIP的去除贡献略有提升,但未达到差异性显著水平。水解作用为OTC和CIP在湿地中去除的次要途径,其对两者去除的平均贡献比例>10.62%;生物作用程度则最小,最大相对平均贡献比例仅为4.39%。然而,污水中传统污染物对2种抗生素水解去除作用和生物去除作用的影响截然相反,传统污染物存在可提升生物作用对OTC和CIP去除的相对贡献比例,但会降低湿地内部OTC和CIP的水解作用贡献比例。这种现象对于OTC来说尤其明显,OTC在处理组1中的水解作用相对平均贡献比例为28.60%,而在处理组2中已降低到21.17%。在SMZ方面,填料吸附作用仍为该类抗生素的主要去除途径,在有、无传统污染物存在下吸附作用的相对平均贡献比例分别为61.61%和74.03%,显著高于其他去除途径。水解作用在SMZ去除过程中可保持稳定的贡献程度,其在有、无传统污染物存在下的相对平均贡献比例分别为12.80%和14.93%,该去除作用受传统污染物的影响较弱。与OTC和CIP不同,SMZ生物去除作用比例显著提升,特别是在有传统污染物存在情况下其相对平均贡献比例可达25.59%。此外,分析数据表明,传统污染物存在与否还会影响生物作用中植物和微生物对SMZ去除的相对贡献比例,传统污染物存在导致微生物作用,对SMZ去除贡献从1.61%显著提升至22.76%,而植物作用则从9.23%降低到2.83%。
3 讨 论
本文围绕人工湿地中水解作用、吸附作用和生物作用,对畜禽养殖污水所含典型抗生素去除贡献情况开展了研究,不同抗生素去除途径定量化分析结果表明,吸附作用为OTC、CIP和SMZ在人工湿地中的首要去除方式,而水解作用和生物作用则表现出相对较低的减量化效果,该结果符合已有文献报道所得出的抗生素在人工湿地中的减量化过程特征[3,8,9]。
兽用抗生素因其两性分子结构属性,其在不同介质(如活性污泥和土壤)中较强吸附特征已被广泛证明[10,11],从而吸附作用成为OTC、CIP和SMZ在人工湿地中的主要去除途径。而OTC和CIP相比,SMZ所展现出的较高吸附去除比例也与它们的物化结构属性差异相关,OTC和CIP具有较多的极性/离子型官能团,这使得此2种抗生素可通过离子交换、阳离子架桥或表面络合等机制与湿地填料形成更为稳定的吸附关系[12,13]。畜禽养殖污水中氮磷营养盐通常具有较高的残留浓度,这一因素会导致出现氮磷营养盐和抗生素之间吸附点位竞争现象,并强化填料吸附固定的抗生素解析过程[14,15]。然而,本实验结果指出,传统污染物的输入虽影响了吸附作用对OTC和SMZ的相对平均贡献比例,但从平均绝对去除量方面比较,其对2种抗生素吸附行为的影响并不明显。分析原因为:1)本实验所选填料来自长期处理畜禽养殖污水的人工湿地中试平台,填料处于氮磷吸附饱和状态,从而降低了氮磷营养盐与抗生素的竞争行为;2)污水中有机物的存在可显著降低抗生素在溶液中的移动行为,强化吸附固定化效果。Zhao等[16]在研究四环素类抗生素与蒙脱石过程中发现,有机质浓度与抗生素吸附量呈正相关,尤其是在酸性和中性条件下该关系更为明显。因此,从长期运维角度分析,传统污染物存在不会对抗生素吸附产生显著影响。
表1 湿地抗生素不同去除途径的相对平均贡献比例
Table 1 The relative average contribution ratio of antibiotic by different removal pathways in wetland %

去除途径处理组1处理组2OTCCIPSMZOTCCIPSMZ水解作用28.60±4.7215.48±2.3614.93±3.4921.17±3.0010.62±1.0712.80±1.97吸附作用70.11±5.8983.08±3.4174.03±6.3474.61±5.9684.98±2.8261.61±8.54生物作用1.29±1.221.44±1.39—4.22±3.124.39±3.18—微生物作用——1.61±0.65——22.76±8.08植物作用——9.23±3.00——2.83±1.98
水解是指化学物质被水解离的作用过程,而药物水解主要是亲电碳原子与离去基团的化学反应行为[17]。实验结果表明,相比生物作用,水解作用为OTC和CIP在人工湿地中的主要降解途径,但该作用效果会受到传统污染物的抑制影响,尤其是OTC的变化响应最为显著。分析原因,这与处理组1、2中溶液pH水平差异存在关系,处理组2中水环境受传统污染物介入影响,其pH水平为7.18,低于处理组1的8.02。Doi等[18]研究发现,酸性条件有助于OTC的稳定,碱性条件则会提升OTC在水环境中的降解速率,这一现象主要归因于水解形成的OH-对土霉素的催化分解反应要强于H+。此外,抗生素稳定性研究表明,OTC结构由不稳定的C及B环和二元酮组成,而SMZ水解所涉及的亲核酰基取代反应和芳香亲核取代反应过程中离去基团在中性条件下具有较强的稳定性[19,20]。因此,人工湿地水解途径对OTC的作用程度要明显高于SMZ,该结果也表明,通过人工湿地吸附作用和水解作用的优化耦合可实现对污水中抗生素的低成本高效去除,但这种机制取决于抗生素的物化特征。
人工湿地中生物作用对3种典型兽用抗生素均可起到降解去除作用,但其对SMZ的作用程度要更显著,这与3类抗生素在活性污泥系统中的行为特征相一致[7,11],但传统污染物存在与否会影响生物作用中植物和微生物对SMZ去除的贡献程度。植物可通过主动传递和蒸腾作用吸收污水中抗生素成分,其中主动传递途径作用强度和抗生素辛醇-水分配系数之间存在密切相关性,极性疏水或者极性亲水的有机物不易被植物吸收,而蒸腾作用强度则与环境气温呈正相关[21,22]。本实验过程所涉及目标抗生素的辛醇-水分配系数表明,OTC、CIP和SMZ难以被植物主动吸收与传递,各系统水量损失程度的相似性也暗示蒸腾作用所带来的被动吸收也不会明显发生在本实验过程中,因此植物吸收作用应不是OTC、CIP和SMZ在人工湿地中的关键途径,而处理组1中植物对SMZ的较高去除贡献率应是由根系微生物和根系分泌物协同作用形成的共代谢机制所致[23]。在芘类有机物降解研究方面,芦苇已被证实可通过根系细菌和碳类分泌物协同作用促进该类污染物的生物降解过程[24]。同时,比较2个处理组中微生物作用也可发现,处理组1中SMZ微生物去除作用贡献比例(2.06%)在第1运行周期内已远远小于处理组2(15.46%)。Oliveira等[25]在研究序批式反应器内厌氧颗粒污泥降解SMZ时也发现,有机物的介入会显著提升SMZ的去除效率。该结果表明,传统污染物介入会立即影响微生物对SMZ的生物降解过程,而非后续生物量增长所导致的降解效率差异,这亦可为前述植物作用推论提供依据。因此,湿地微生物对抗生素的降解应为人工湿地中生物作用的主要体现形式。
4 结 论
人工湿地中水解作用、吸附作用和生物作用在抗生素去除过程中皆有贡献效果,其中吸附作用的贡献程度和稳定性能最高。填料吸附和水解降解的耦合作用为污水中土霉素和环丙沙星在人工湿地中的去除机制模式,而磺胺二甲基嘧啶去除则主要依靠填料吸附和微生物降解的协同作用。从长期运维角度分析,传统污染物存在不会对抗生素吸附产生显著的作用影响,但其会抑制土霉素和环丙沙星的水解过程及提升磺胺二甲基嘧啶的微生物降解效果。
[1] ZHOU L J, YING G G, LIU S, et al. Excretion masses and environmental occurrence of antibiotics in typical swine and dairy cattle farms in China[J]. Science of the Total Environment, 2013, 444(2):183-195.
[2] 傅海霞, 刘怡, 董志英,等.抗生素与重金属复合污染的生态毒理效应研究进展[J]. 环境工程, 2016, 34(4):60-63,104.
[3] LIU L, LIU Y H, WANG Z, et al. Behavior of tetracycline and sulfamethazine with corresponding resistance genes from swine wastewater in pilot-scale constructed wetlands[J]. Journal of Hazardous Materials, 2014, 278:304-310.
[4] 张鹏飞, 刘晓文, 李杰,等. 养殖废水中抗生素去除处理工艺的研究现状[J]. 净水技术, 2018, 37(4):60-65.
[5] 程宪伟, 梁银秀, 祝惠,等. 人工湿地处理水体中抗生素的研究进展[J]. 湿地科学, 2017, 15(1):128-134.
[6] ZHANG Q Q, YING G G, PAN C G, et al. Comprehensive evaluation of antibiotics emission and fate in the river basins of China: source analysis, multimedia modeling, and linkage to bacterial resistance[J]. Environmental Science & Technology, 2015, 49(11): 6772-6782.
[7] LIAO J, LIU C X, LIU L, et al. Influence of hydraulic retention time on behavior of antibiotics and antibiotic resistance genes in aerobic granular reactor treating biogas slurry[J]. Frontiers of Environmental Science & Engineering, 2019, 13(3):59-67.
[8] LIU L, LI J, FAN H Y, et al. Fate of antibiotics from swine wastewater in constructed wetlands with different flow configurations[J]. International Biodeterioration & Biodegradation, 2019, 140: 119-125.
[9] HIJOSA-VALSERO M, FINK G, SCHLÜSENER M P, et al. Removal of antibiotics from urban wastewater by constructed wetland optimization[J]. Chemosphere, 2011, 83(5): 713-719.
[10] DAGHRIR R, DROGUI P. Tetracycline antibiotics in the environment: a review[J]. Environmental Chemistry Letters, 2013, 11(3): 209-227.
[11] LI B, ZHANG T. Biodegradation and adsorption of antibiotics in the activated sludge process[J]. Environmental Science & Technology, 2010, 44(9): 3468-3473.
[12] SONG X C, LIU D F, ZHANG G W, et al. Adsorption mechanisms and the effect of oxytetracycline on activated sludge[J]. Bioresource Technology, 2014, 151: 428-431.
[13] WU Q F, LI Z H, HONG H L, et al. Adsorption and intercalation of ciprofloxacin on montmorillonite[J]. Applied Clay Science, 2010, 50(2): 204-211.
[14] CHENG D M, FENG Y, LIU Y W, et al. Quantitative models for predicting adsorption of oxytetracycline, ciprofloxacin and sulfamerazine to swine manures with contrasting properties[J]. Science of the Total Environment, 2018, 634: 1148-1156.
[15] LI J, ZHANG H, YUAN G D. Phosphate affects adsorption and desorption of oxytetracycline in the seawater-sediment systems[J]. Environmental Science and Pollution Research, 2018, 25(28): 28160-28168.
[16] ZHAO Y P, GU X Y, GAO S X, et al. Adsorption of tetracycline (TC) onto montmorillonite: cations and humic acid effects[J]. Geoderma, 2012, 183/184: 12-18.
[17] WATERMAN K C, ADAMI R C, ALSANTE K M, et al. Hydrolysis in pharmaceutical formulations[J]. Pharmaceutical Development and Technology, 2002, 7(2): 113-146.
[18] DOI A M, STOSKOPF M K. The kinetics of oxytetracycline degradation in deionized water under varying temperature, pH, light, substrate, and organic matter[J]. Journal of Aquatic Animal Health, 2000, 12(3): 246-253.
![]() A, STOLTE S, MATZKE M, et al. Hydrolysis of sulphonamides in aqueous solutions[J]. Journal of Hazardous Materials, 2012, 221/222: 264-274.
A, STOLTE S, MATZKE M, et al. Hydrolysis of sulphonamides in aqueous solutions[J]. Journal of Hazardous Materials, 2012, 221/222: 264-274.
[20] XUAN R C, ARISI L, WANG Q Q, et al. Hydrolysis and photolysis of oxytetracycline in aqueous solution[J]. Journal of Environmental Science and Health Part B, 2009, 45(1): 73-81.
[21] LIU L, LIU Y H, LIU C X, et al. Potential effect and accumulation of veterinary antibiotics in Phragmites australis under hydroponic conditions[J]. Ecological Engineering, 2013, 53: 138-143.
[22] DETTENMAIER E M, DOUCETTE W J, BUGBEE B. Chemical hydrophobicity and uptake by plant roots[J]. Environmental Science & Technology, 2008, 43(2): 324-329.
[23] DALTON H, STIRLING D I. Co-metabolism[J]. Philosophical Transactions of the Royal Society of London. Series B, Biological Sciences, 1982, 297(1088): 481-496.
[24] TOYAMA T, FURUKAWA T, MAEDA N, et al. Accelerated biodegradation of pyrene and benzo [a] pyrene in the Phragmites australis rhizosphere by bacteria-root exudate interactions[J]. Water Research, 2011, 45(4): 1629-1638.
[25] OLIVEIRA G H D, SANTOS-NETO A J, ZAIAT M. Evaluation of sulfamethazine sorption and biodegradation by anaerobic granular sludge using batch experiments[J]. Bioprocess and Biosystems Engineering, 2016, 39(1): 115-124.